Analysis of multi-organ full-length transcriptome and structural genes involved in flavonoid biosynthesis of Gymnosphaera podophylla Dalla Torre & Sarnth.
-
摘要:
黑桫椤(Gymnosphaera podophylla Dalla Torre & Sarnth.)为著名的孑遗蕨类,具有很强的环境适应能力,然而其适应性机制尚不清楚。本研究采用PacBio和Illumina技术对黑桫椤的根、羽轴和羽片进行转录组测序,分别生成12 879、14 185和16 084个全长unigenes。基因表达分析结果表明,与黑桫椤抗干旱、缺水胁迫和生物胁迫相关的基因表达水平较高。根、羽轴和羽片特异性上调基因均显著富集到KEGG代谢通路中的“苯丙烷生物合成途径”,根和羽轴的器官特异性上调基因还显著富集到“类黄酮生物合成途径”。共有192个全长unigenes被注释为类黄酮生物合成途径所涉及的13个酶结构基因,其中包括112个差异表达基因(DEGs),表明黑桫椤类黄酮生物合成途径较为保守,且存在器官特异性差异表达基因。本文对黑桫椤多器官全长转录组和类黄酮生物合成途径结构基因进行了综合分析,为进一步研究其对环境的适应性提供了丰富的遗传资源。
Abstract:Gymnosphaera podophylla Dalla Torre & Sarnth. is a famous relict tree fern with strong environmental adaptability. However, the mechanisms underlying its adaptability remain unclear. In this study, the PacBio and Illumina platforms were used to sequence the root, rachis, and pinna transcriptomes of G. podophylla, resulting in the generation of 12 879, 14 185, and 16 084 full-length unigenes, respectively. Transcript quantification showed that these unigenes were related to drought resistance and biological stress and were highly expressed. KEGG enrichment analysis indicated that the up-regulated genes in the roots, rachis, and pinna were enriched in the "phenylpropane biosynthesis pathway", while the up-regulated genes in the roots and rachis were enriched in the "flavonoid biosynthetic pathway". A total of 192 full-length unigenes were annotated as structural genes involving 13 enzymes in the flavonoid biosynthesis pathway, including 112 differentially expressed genes (DEGs), suggesting that the flavonoid biosynthesis pathway was conserved in G. podophylla, with organ-specific DEGs. This research is the first to perform a comprehensive analysis of the full-length transcriptome across multiple organs in G. podophylla and to investigate the structural genes of the flavonoid biosynthetic pathway. This study provides an abundance of genetic resources for further examination of environmental adaptation in this species.
-
VQ(Valine-glutamine)是植物特有的一类蛋白,因其包含一个特有的保守基序FxxxVQxLTG而得名(F:苯丙氨酸,V:缬氨酸,Q:谷氨酰胺,L:亮氨酸,T:苏氨酸,G:甘氨酸,x 代表任意氨基酸)[1-3]。首个VQ蛋白SIB1(SIGMA FACTOR-BINDING PROTEIN1)在拟南芥(Arabidopsis thaliana (L.) Heynh.)中被发现[4],随后,从拟南芥中又陆续鉴定出33个VQ蛋白。目前,科研人员已在茶(Camellia sinensis (L.) O. Ktze. )[2]、大豆(Glycine max (L.) Merr.)[5]、小麦(Triticum aestivum L.)[6]及烟草(Nicotiana tabacum L.)[3]中分别鉴定出25、75、113和59个VQ基因。
VQ蛋白在植物应答生物与非生物胁迫中发挥着重要作用[7, 8],其作用机制主要有两种。一是通过与WRKY转录因子相互作用发挥功能,如AtVQ23和AtVQ16通过与WRKY33的WRKY结构域结合,增强后者与靶基因的结合能力,从而影响植物的抗病能力[9]。而AtVQ9与WRKY8互作抑制了WRKY8与W-box的结合活性,从而负调控植物的抗盐性[10]。在番茄(Solanum lycopersicum L.)叶片中,SlVQ7与SlWRKY37互作可提高后者的稳定性,并转录激活其下游靶基因,从而正调控茉莉酸及黑暗诱导的叶片衰老[11]。二是作为丝裂原活化蛋白激酶(MAPK)家族的磷酸化底物发挥作用[12-14]。如拟南芥MKS1(AtVQ21)作为AtMPK4的底物,与WRKY25和WRKY33相互作用,有助于激活MPK4调节的病原体防御反应[3]。
多年生黑麦草(Lolium perenne L.)为禾本科早熟禾亚科黑麦草属植物,是世界上广泛种植的冷季型草坪草及牧草[15],具有分蘖多、成坪速度快、粗蛋白含量高、耐牧性好等优点。多年生黑麦草在生长发育过程中常面临高温、盐碱、荫蔽等非生物胁迫,严重影响草坪质量和牧草品质。因此,鉴定多年生黑麦草VQ基因,探究其在应答非生物胁迫中的作用十分必要。然而,目前尚未见相关报道。本研究系统鉴定了多年生黑麦草VQ蛋白家族成员,并对其保守结构域、进化关系、共线性、亚细胞定位及其在高温、盐碱、黑暗等非生物胁迫下的表达模式进行了分析,研究结果旨在为进一步探讨VQ蛋白在多年生黑麦草生长发育及应答非生物逆境胁迫中的作用奠定基础。
1. 材料与方法
1.1 实验材料
本研究以多年生黑麦草品种‘百灵鸟’为实验材料。黑麦草种子于营养土(基质∶蛭石=1∶1)中萌发,萌发后转移至光照培养箱,光周期为16 h / 8 h(光 / 黑暗),温度为25 ℃ / 22 ℃(光 / 黑暗)。待植株生长1个月后进行黑暗、高温和NaCl处理。黑暗处理的条件为:25 ℃ / 22 ℃,24 h黑暗,分别于处理0、2、8 d后取样。高温处理的条件为:42 ℃,光周期16 h / 8 h,分别于处理0、1、24 h后取样。NaCl处理的条件为:25 ℃ / 22 ℃,光周期16 h / 8 h,使用200 mL 200 mmol/L的NaCl溶液进行浇灌,分别于盐处理0、3、38 h后取样。
1.2 多年生黑麦草VQ基因家族成员的鉴定
以Pfam数据库中VQ结构域(PF05678)的隐马尔可夫模型(HMM)图谱作为查询,通过HMMER搜索多年生黑麦草基因组,鉴定了可能的VQ蛋白序列。使用Pfam数据库(http://pfam.xfam.org/)进一步确认了这些序列。在拟南芥基因组数据库(TAIR)中下载拟南芥全基因组序列及其注释信息。
1.3 多年生黑麦草VQ家族成员的理化性质及亚细胞定位
使用在线网站(https://web.expasy.org/compute_pi/)分析多年生黑麦草VQ蛋白的理化性质。使用BIOINF(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-)在线工具对多年生黑麦草VQ家族成员进行亚细胞定位预测。
1.4 多年生黑麦草VQ家族基因系统发育树构建
使用MEGAX(http://www.megasoftware.net)软件的ML法构建系统发育树,进行系统发育分析,并用iTOL(https://itol.embl.de/)网站进行美化。
1.5 多年生黑麦草VQ保守基序及基因结构分析
利用MEME(https://meme-suite.org/meme/tools/meme)网站对VQ蛋白的氨基酸序列进行保守基序分析。结合植物基因组序列和注释文件,用TBtools提取VQ基因的基因组序列及CDS序列,用TBtools软件工具中的Gene Structure View进行可视化。
1.6 VQ基因染色体分布及种内共线性分析
结合植物基因组序列和注释文件,使用TBtools软件工具中的Amazing Gene Location From GFF3/GTF File进行可视化,使用TBtools软件工具中的one step MCScanX进行种内基因组比对,并用Multiple Synreny Plot进行可视化。
1.7 胁迫条件下多年生黑麦草VQ基因的表达模式分析
利用Trizol试剂(Takara,大连)提取多年生黑麦草在不同非生物胁迫处理后的总RNA。使用反转录试剂盒Hifair® Ⅲ 1st Strand cDNA Synthesis SuperMix for qPCR(gDNA digester plus)(Yeasen,上海)合成cDNA。利用Hieff® qPCR SYBR Green Master Mix(High Rox Plus)试剂盒(Yeasen,上海)在Step One Plus Real-Time PCR Systems系统上进行qRT-PCR反应。反应体系总体积为20 μL,包括2 μL cDNA,上、下游引物各0.8 μL,10 μL SYBR,用ddH2O补足体积。反应程序为:95 ℃预变性5 min;95 ℃变性10 s,60 ℃退火20 s,72 ℃延伸20 s,40个循环,系统自带熔解曲线。引物序列见附表1
1 ,由擎科公司(擎科,武汉)合成,多年生黑麦草eEF1A(s)为内参基因。采用2−△△Ct方法计算候选基因的相对表达量,实验设3次重复。利用TBtools绘制热图,进行表达模式分析。2. 结果与分析
2.1 LpVQ家族成员鉴定
为了系统分析多年生黑麦草的VQ基因,以VQ结构域(PF05678)为参考,从全基因组水平挖掘其VQ家族基因。结果显示,多年生黑麦草中共存在52个VQ基因。对这些基因的编码产物LpVQ家族成员进行理化性质分析,发现LpVQs的氨基酸长度介于118~427 aa,蛋白分子量范围为11.67~43.84 kD,等电点在5.01~11.6。通过BIOINF在线网站对LpVQ家族成员进行亚细胞定位预测,发现48个蛋白定位在细胞核中,LpVQ22为叶绿体与细胞核共定位,LpVQ33为细胞壁与细胞核共定位,LpVQ37为细胞膜与细胞核共定位,而LpVQ45预测定位在细胞膜中(附表2
1 )。2.2 LpVQ家族系统进化树分析
为了分析LpVQs的进化关系,利用多年生黑麦草的52个LpVQ蛋白和拟南芥的34个AtVQ蛋白共同构建系统进化树。结果显示,拟南芥与多年生黑麦草的VQ家族成员可划分为7个亚家族(Ⅰ~Ⅶ),分别包含5、8、6、11、12、6和4个LpVQ蛋白。亚家族Ⅰ中只包含LpVQs,亚家族Ⅳ、Ⅴ和Ⅵ中含有较多的LpVQ家族成员,而AtVQ家族成员则更多地归类于亚家族Ⅱ、Ⅲ和Ⅶ(图1)。进一步分析发现,多年生黑麦草与拟南芥的VQ家族间存在4对直系同源基因,分别是LpVQ6 / AtVQ7、LpVQ11 / AtVQ9、LpVQ50 / AtVQ32和LpVQ5 / AtVQ31。
2.3 LpVQ家族成员保守基序和基因结构分析
为了研究蛋白的结构多样性,利用MEME在线软件对52个LpVQ蛋白的保守基序进行分析,结果发现17%的LpVQs含有1个Motif,48%的LpVQs含有两个Motif,19%的LpVQs含有3个Motif,6%的LpVQs含有4个Motif,10%的LpVQs含有5个Motif。所有家族成员均具有Motif1,表明它是LpVQ家族的核心保守结构域(图2)。
此外,本研究用TBtools工具对LpVQs进行了基因结构分析。结果发现,46个LpVQ基因没有内含子,5个LpVQ基因含有1个内含子(LpVQ24、LpVQ29、LpVQ31、LpVQ32、LpVQ52),仅有1个基因(LpVQ161)含有两个内含子(附图1
1 )。2.4 LpVQ家族成员染色体定位以及共线性分析
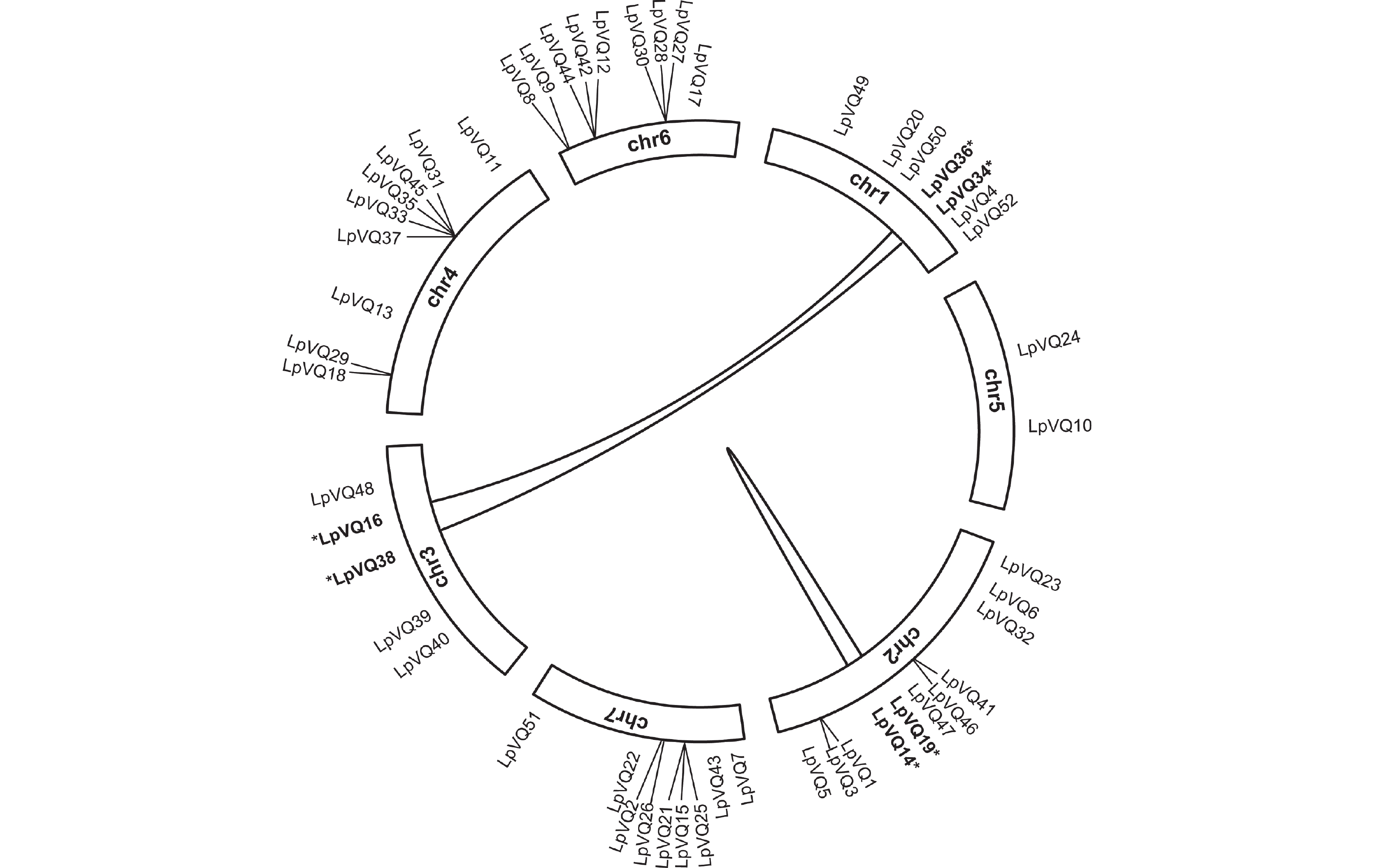
染色体定位分析结果表明,LpVQ基因分布在多年生黑麦草的7条染色体上,其中分布在chr1上的有7个,占13%,分布在chr2上的最多,为11个,占21%,分布在chr3上的为5个,占10%,分布在chr4、chr6和chr7上的基因均各有9个,各占17%,而分布在chr5上的最少,仅有2个,占4%(图3)。
为了了解LpVQ基因之间的进化关系,本研究对VQ家族成员进行了种内共线性分析。结果表明,3对VQ基因之间存在共同进化关系,分别是LpVQ16与LpVQ36、LpVQ34与LpVQ38以及LpVQ14与LpVQ19(图4)。
2.5 LpVQ基因响应非生物胁迫的表达模式分析
从7个亚家族中分别随机选取3个LpVQ基因,检测其在黑暗、高温以及盐胁迫下多年生黑麦草中的表达量变化。结果显示,黑暗处理后,只有4个LpVQ基因的表达持续上调。大部分LpVQ基因的表达量在黑暗处理2 d后下降,其中,LpVQ2、LpVQ8、LpVQ14、LpVQ22及LpVQ38的表达量在黑暗处理8 d后又显著上升(图5:A)。
![]() 图 5 LpVQ基因响应黑暗、高温及NaCl胁迫的表达模式分析A:黑暗处理;B:高温处理;C:NaCl处理。红色、蓝色和黄色分别代表上调、下调和没有表达差异的基因。Figure 5. Expression analyses of LpVQs under dark, high temperature and NaCl treatmentsA: Dark treatment; B: Heat treatment; C: NaCl treatment. Red, blue, and yellow represent up-regulated, down-regulated, and no differential genes, respectively.
图 5 LpVQ基因响应黑暗、高温及NaCl胁迫的表达模式分析A:黑暗处理;B:高温处理;C:NaCl处理。红色、蓝色和黄色分别代表上调、下调和没有表达差异的基因。Figure 5. Expression analyses of LpVQs under dark, high temperature and NaCl treatmentsA: Dark treatment; B: Heat treatment; C: NaCl treatment. Red, blue, and yellow represent up-regulated, down-regulated, and no differential genes, respectively.42 ℃高温处理后,LpVQ的表达模式被分为3类。LpVQ18、LpVQ8、LpVQ28、LpVQ2、LpVQ7、LpVQ4及LpVQ12受1 h高温处理的显著诱导,而高温处理24 h后,其表达又显著下调。LpVQ9、LpVQ22、LpVQ15、LpVQ3、LpVQ42、LpVQ6、LpVQ10、LpVQ1、LpVQ38的表达量在1 h和24 h高温处理后均显著下降。LpVQ5、LpVQ27、LpVQ34、LpVQ14和LpVQ44的表达量只在24 h高温处理后下降(图5:B)。
NaCl处理3 h后,13个LpVQ基因显著上调表达,其中LpVQ4的表达上升了13倍。而NaCl处理38 h后,大部分基因的表达明显下调。仅有3个基因的表达受盐诱导持续上调,分别是 LpVQ8、LpVQ10和LpVQ38(图5:C)。
3. 讨论
近年来,VQ基因已经在多种植物中被发掘鉴定,包括拟南芥、水稻(Oryza sativa L.)、大豆和葡萄(Vitis vinifera L.)等[16-19],而关于多年生黑麦草VQ家族基因的鉴定和分析还鲜见报道。本研究通过全基因组鉴定,得到52个多年生黑麦草LpVQ基因,并系统分析了其理化特性、蛋白结构、基因结构、共线性、进化关系以及响应逆境胁迫的表达模式。
基因结构分析发现,88.46%的LpVQ基因无内含子,与之类似,在拟南芥[16]、水稻[17]、玉米(Zea mays L.)[20]和马铃薯(Solanum tuberosum L.)[21]中,无内含子的VQ基因分别占88.2%、92.5%、88.5%和91.89%。而苔藓(Bryophyta)[22]中无内含子的VQ基因比例则远低于以上物种,仅为28.0%,说明VQ基因可能在长期进化过程中丢失了内含子。
系统进化分析结果显示,多年生黑麦草和拟南芥的VQ家族成员可被分为7个亚家族,且LpVQs与AtVQs亲缘关系较远,只存在4对直系同源基因,分别是LpVQ6 / AtVQ7、LpVQ11 / AtVQ9、LpVQ50 / AtVQ32和LpVQ5 / AtVQ31。根据拟南芥VQ基因的功能,可以推测多年生黑麦草中其直系同源VQ基因的功能。有研究指出,AtVQ9作为AtWRKY8的抑制子,能提高拟南芥的耐盐性[9],由此推测,其直系同源基因LpVQ11可能也在多年生黑麦草抵御盐胁迫中发挥重要作用。
VQ基因在植物响应非生物胁迫中发挥着重要作用。如,过表达PtVQ1基因显著增强了转基因拟南芥的耐盐性[23];而过表达MdVQ37则提高了转基因苹果(Malus pumila Mill.)对高温胁迫的敏感性[24]。本研究对黑暗、高温及盐胁迫处理下多年生黑麦草的VQ表达量进行了分析。结果显示,LpVQ基因广泛响应黑暗、高温及盐胁迫,且大部分LpVQ基因均在胁迫初期即做出快速响应,这与西葫芦(Cucurbita pepo L.)[8]、黄瓜(Cucumis sativus L.)[25]等物种中的研究结果相类似。然而,LpVQ基因的表达模式在不同胁迫处理下也存在差异。如,LpVQ1在黑暗胁迫处理后持续下调表达,而在高温和盐胁迫下持续上调表达,推测该基因可能在不同胁迫中发挥着不同的作用。尽管LpVQ可能参与多年生黑麦草对黑暗、高温及盐等胁迫响应的调控,但其具体功能还需要进一步验证。
综上所述,本研究对多年生黑麦草VQ基因家族成员进行了系统鉴定与分析,为后续验证VQ家族成员在多年生黑麦草应答生物或非生物胁迫中的功能提供了基础。
1 如需查阅附表内容请登录《植物科学学报》网站(http://www.plantscience.cn)查看本期文章。2 1~7)如需查阅附件内容请登录《植物科学学报》网站(http://www.plantscience.cn)查看本期文章。 -
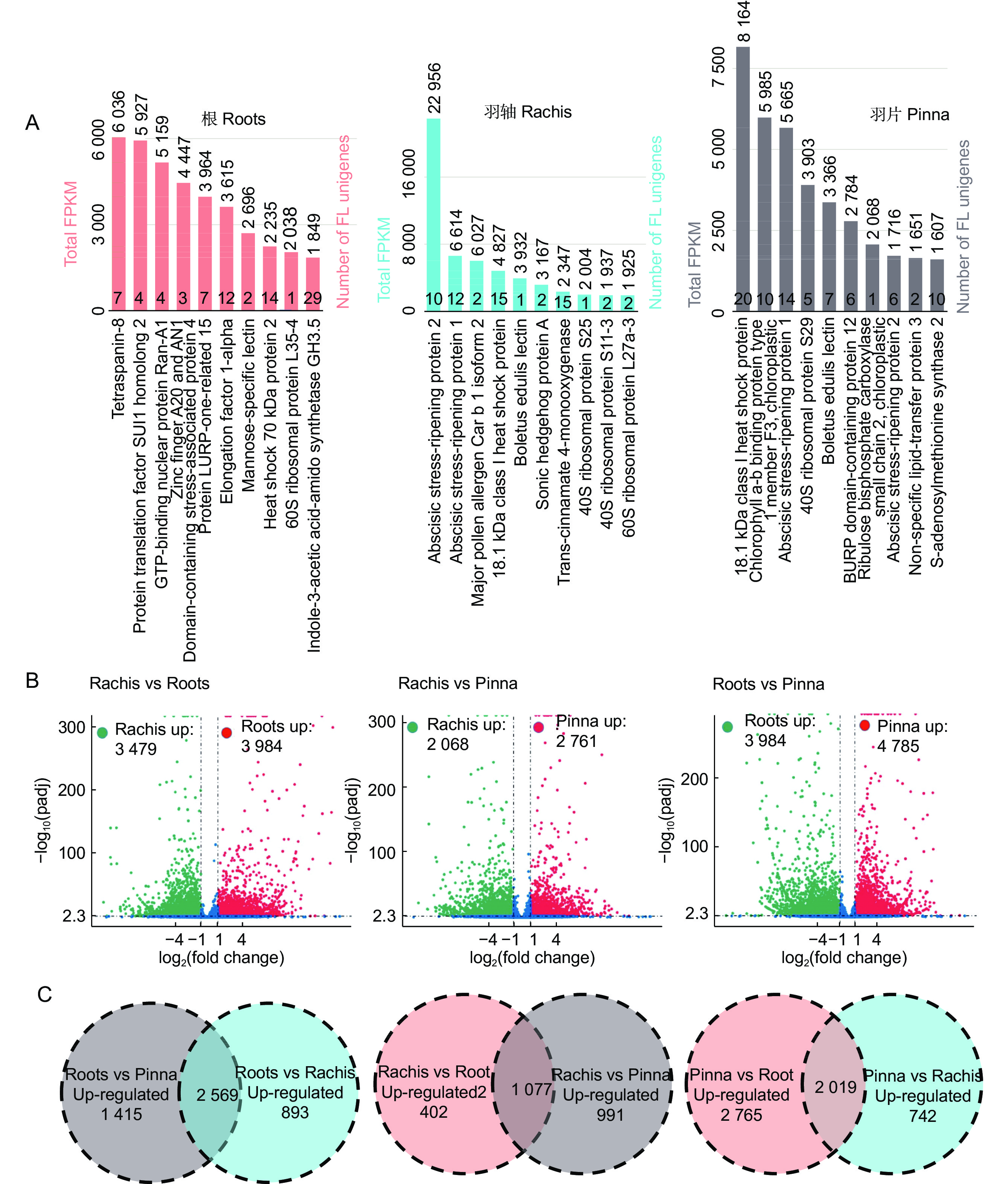
图 1 黑桫椤3种器官的高表达和差异表达基因
A:各器官前10个高表达基因。柱状图底部的数字代表注释为同一蛋白的FL unigenes数目。柱状图上方的数字代表注释为同一蛋白的FL unigenes的总FPKM值;B:从左至右依次为根和羽轴、羽片和羽轴、羽片和根的成对比较的差异表达基因数量;C:从左至右依次是根、羽轴、羽片相对于其他两种器官上调的基因数量。
Figure 1. Highly expressed genes and DEGs in three organs of Gymnosphaera podophylla
A: The top ten highly expressed genes in three organs. Number of FL unigenes annotated as same protein is shown at the bottom of the bar. Sum of FPKM values of FL unigenes annotated for the same protein are above the bar; B: From left to right is the number of DEGs in roots versus rachis, pinna versus rachis, and pinna versus roots, respectively; C: From left to right, number of genes up-regulated in roots, rachis, and pinna relative to the other two organs, respectively.
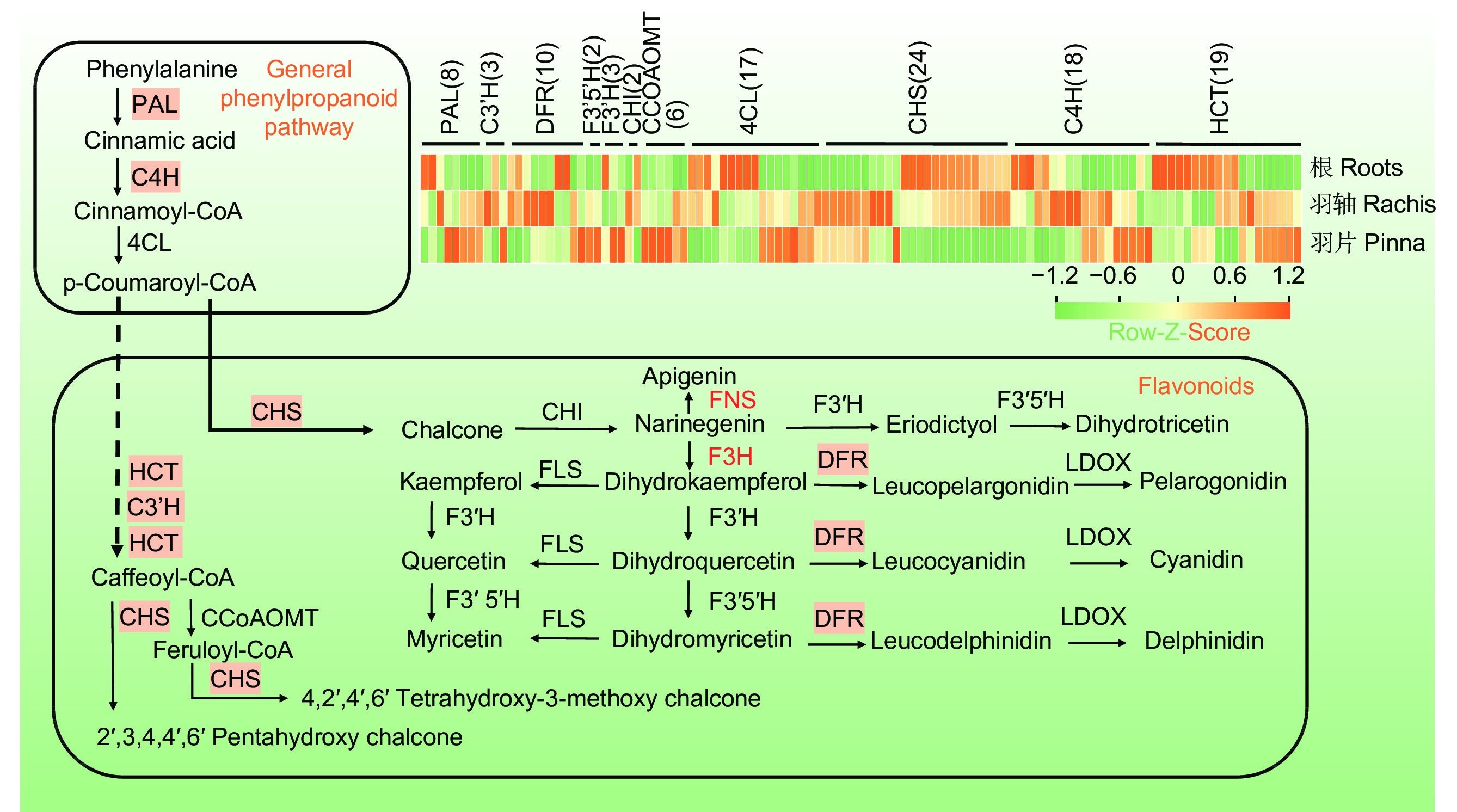
图 2 类黄酮生物合成通路
热图展示了3种器官中类黄酮生物合成途径的DEG表达情况。高亮表示该酶有多个FL unigenes(>10),标红字体表示未发现FNS和F3H的FL unigenes,虚线箭头表示由多个酶参与的多步反应。
Figure 2. Flavonoid biosynthesis pathway
Heatmap showing DEG expression profiles of flavonoid biosynthesis pathway in three organs. Highlighted gene contains multiple unigenes (>10) in G. podophylla. Red font indicates F3H and FNS genes were not found in transcriptome. Dotted arrows indicate multi-step reaction with multiple enzymes.
表 1 全长转录组和组装数据
Table 1 Full-length transcriptome and assembled data
器官
Organ基因类型
Unigenes type碱基量
Raw reads / Gb总数
Total numberGC含量
GC content / %N50 / bp 根 组装 8.20 35 921 50.00 1838 全长 16.26 12 879 48.00 1979 羽轴 组装 8.81 30 752 49.70 1938 全长 18.39 14 185 47.00 1898 羽片 组装 6.74 29 938 49.30 1804 全长 21.99 16 048 47.00 1875 -
[1] 苏应娟,王艇,郑博,江宇,欧阳蒲月,等. 根据cpDNA trnL-F非编码区序列变异分析黑桫椤海南和广东种群的遗传结构与系统地理[J]. 生态学报,2004,24(5):914−919. doi: 10.3321/j.issn:1000-0933.2004.05.008 Su YJ,Wang T,Zheng B,Jiang Y,Ouyang PY,et al. Genetic structure and phylogeography of populations of Alsophila podophylla in Hainan and Guangdong,southern China,based on cpDNA trnL-F noncoding sequences[J]. Acta Ecologica Sinica,2004,24(5):914−919. doi: 10.3321/j.issn:1000-0933.2004.05.008
[2] Ppg I. A community-derived classification for extant lycophytes and ferns[J]. J Syst Evol,2016,54(6):563−603. doi: 10.1111/jse.12229
[3] 谢春平,赵柏松,刘大伟,方彦. 霸王岭自然保护区黑桫椤种群结构特征分析[J]. 四川农业大学学报,2018,36(6):765−771. Xie CP,Zhao BS,Liu DW,Fang Y. Study on the population structure of Alsophila podophylla Hook. in Bawangling nature reserve[J]. Journal of Sichuan Agricultural University,2018,36(6):765−771.
[4] 赵则海. 黑桫椤叶中总黄酮含量的测定[J]. 肇庆学院学报,2014,35(5):42−44. doi: 10.3969/j.issn.1009-8445.2014.05.011 Zhao ZH. Determination of flavonoids contents in leaves of Alsophila podophylla[J]. Journal of Zhaoqing University,2014,35(5):42−44. doi: 10.3969/j.issn.1009-8445.2014.05.011
[5] Dong NQ,Lin HX. Contribution of phenylpropanoid metabolism to plant development and plant-environment interactions[J]. J Integr Plant Biol,2021,63(1):180−209. doi: 10.1111/jipb.13054
[6] 葛诗蓓,张学宁,韩文炎,李青云,李鑫. 植物类黄酮的生物合成及其抗逆作用机制研究进展[J]. 园艺学报,2023,50(1):209−224. Ge SB,Zhang XN,Han WY,Li QY,Li X. Research progress on plant flavonoids biosynthesis and their anti-stress mechanism[J]. Acta Horticulturae Sinica,2023,50(1):209−224.
[7] Zhang XX,Wang X,Wang ML,Cao JG,Xiao JB,Wang QX. Effects of different pretreatments on flavonoids and antioxidant activity of Dryopteris erythrosora leave[J]. PLoS One,2019,14(1):e0200174. doi: 10.1371/journal.pone.0200174
[8] Rybczyński JJ,Marczak Ł,Stobiecki M,Strugała A,Mikuła A. The metabolite content of the post-culture medium of the tree fern Cyathea delgadii Sternb. cell suspension cultured in the presence of 2,4-D and BAP[J]. Int J Mol Sci,2022,23(19):11783. doi: 10.3390/ijms231911783
[9] Hong YF,Wang Z,Li MH,Su YJ,Wang T. First multi-organ full-length transcriptome of tree fern Alsophila spinulosa highlights the stress-resistant and light-adapted genes[J]. Front Genet,2022,12:784546. doi: 10.3389/fgene.2021.784546
[10] Sun MY,Li JY,Li D,Huang FJ,Wang D,et al. Full-length transcriptome sequencing and modular organization analysis of the naringin/neoeriocitrin-related gene expression pattern in Drynaria roosii[J]. Plant Cell Physiol,2018,59(7):1398−1414.
[11] Niu M,Fu J,Ni R,Xiong RL,Zhu TT,et al. Functional and structural investigation of chalcone synthases based on integrated metabolomics and transcriptome analysis on flavonoids and anthocyanins biosynthesis of the fern Cyclosorus parasiticus[J]. Front Plant Sci,2021,12:757516. doi: 10.3389/fpls.2021.757516
[12] Bolger AM,Lohse M,Usadel B. Trimmomatic:a flexible trimmer for Illumina sequence data[J]. Bioinformatics,2014,30(15):2114−2120. doi: 10.1093/bioinformatics/btu170
[13] Grabherr MG,Haas BJ,Yassour M,Levin JZ,Thompson DA,et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nat Biotechnol,2011,29(7):644−652. doi: 10.1038/nbt.1883
[14] Davidson NM,Oshlack A. Corset:enabling differential gene expression analysis for de novo assembled transcriptomes[J]. Genome Biol,2014,15(7):410.
[15] Salmela L,Rivals E. LoRDEC:accurate and efficient long read error correction[J]. Bioinformatics,2014,30(24):3506−3514. doi: 10.1093/bioinformatics/btu538
[16] Fu LM,Niu BF,Zhu ZW,Wu ST,Li WZ. CD-HIT:accelerated for clustering the next-generation sequencing data[J]. Bioinformatics,2012,28(23):3150−3152. doi: 10.1093/bioinformatics/bts565
[17] Altschul SF,Gish W,Miller W,Myers EW,Lipman DJ. Basic local alignment search tool[J]. J Mol Biol,1990,215(3):403−410. doi: 10.1016/S0022-2836(05)80360-2
[18] Buchfink B,Xie C,Huson DH. Fast and sensitive protein alignment using DIAMOND[J]. Nat Methods,2015,12(1):59−60. doi: 10.1038/nmeth.3176
[19] Finn RD,Clements J,Eddy SR. HMMER web server:interactive sequence similarity searching[J]. Nucleic Acids Res,2011,39(S2):W29−W37.
[20] Shimizu K,Adachi J,Muraoka Y. ANGLE:a sequencing errors resistant program for predicting protein coding regions in unfinished cDNA[J]. J Bioinform Comput Biol,2006,4(3):649−664. doi: 10.1142/S0219720006002260
[21] Beier S,Thiel T,Münch T,Scholz U,Mascher M. MISA-web:a web server for microsatellite prediction[J]. Bioinformatics,2017,33(16):2583−2585. doi: 10.1093/bioinformatics/btx198
[22] Zheng Y,Jiao C,Sun HH,Rosli HG,Pombo MA,et al. iTAK:a program for genome-wide prediction and classification of plant transcription factors,transcriptional regulators,and protein kinases[J]. Mol Plant,2016,9(12):1667−1670. doi: 10.1016/j.molp.2016.09.014
[23] Sun L,Luo HT,Bu DC,Zhao GG,Yu KT,et al. Utilizing sequence intrinsic composition to classify protein-coding and long non-coding transcripts[J]. Nucleic Acids Res,2013,41(17):e166. doi: 10.1093/nar/gkt646
[24] Kong L,Zhang Y,Ye ZQ,Liu XQ,Zhao SQ,et al. CPC:assess the protein-coding potential of transcripts using sequence features and support vector machine[J]. Nucleic Acids Res,2007,35(S2):W345−W349.
[25] Finn RD,Coggill P,Eberhardt RY,Eddy SR,Mistry J,et al. The Pfam protein families database:towards a more sustainable future[J]. Nucleic Acids Res,2016,44(D1):D279−D285. doi: 10.1093/nar/gkv1344
[26] Li AM,Zhang JY,Zhou ZY. PLEK:a tool for predicting long non-coding RNAs and messenger RNAs based on an improved k-mer scheme[J]. BMC Bioinformatics,2014,15(1):311. doi: 10.1186/1471-2105-15-311
[27] Langmead B,Salzberg SL. Fast gapped-read alignment with Bowtie 2[J]. Nat Methods,2012,9(4):357−359. doi: 10.1038/nmeth.1923
[28] Li B,Dewey CN. RSEM:accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics,2011,12:323. doi: 10.1186/1471-2105-12-323
[29] Anders S,Huber W. Differential expression analysis for sequence count data[J]. Genome Biol,2010,11(10):R106. doi: 10.1186/gb-2010-11-10-r106
[30] Chen CJ,Chen H,Zhang Y,Thomas HR,Frank MH,et al. TBtools:an integrative toolkit developed for interactive analyses of big biological data[J]. Mol Plant,2020,13(8):1194−1202. doi: 10.1016/j.molp.2020.06.009
[31] Guo L,Qi YT,Mu Y,Zhou J,Lu WH,Tian ZD. Potato StLecRK-Ⅳ. 1 negatively regulates late blight resistance by affecting the stability of a positive regulator StTET8[J]. Hortic Res,2022,9:uhac010.
[32] Li JJ,Li Y,Yin ZG,Jiang JH,Zhang MH,et al. OsASR5 enhances drought tolerance through a stomatal closure pathway associated with ABA and H2O2 signalling in rice[J]. Plant Biotechnol J,2017,15(2):183−196. doi: 10.1111/pbi.12601
[33] Rutter BD,Innes RW. Extracellular vesicles isolated from the leaf apoplast carry stress-response proteins[J]. Plant Physiol,2017,173(1):728−741. doi: 10.1104/pp.16.01253
[34] Maskin L,Gudesblat GE,Moreno JE,Carrari FO,Frankel N,et al. Differential expression of the members of the Asr gene family in tomato (Lycopersicon esculentum)[J]. Plant Sci,2001,161(4):739−746. doi: 10.1016/S0168-9452(01)00464-2
[35] Dominguez PG,Conti G,Duffy T,Insani M,Alseekh S,et al. Multiomics analyses reveal the roles of the ASR1 transcription factor in tomato fruits[J]. J Exp Bot,2021,72(18):6490−6509. doi: 10.1093/jxb/erab269
[36] Golan I,Dominguez PG,Konrad Z,Shkolnik-Inbar D,Carrari F,Bar-Zvi D. Tomato ABSCISIC ACID STRESS RIPENING (ASR) gene family revisited[J]. PLoS One,2014,9(10):e107117. doi: 10.1371/journal.pone.0107117
[37] Ritenour MA,Kochhar S,Schrader LE,Hsu TP,Ku MSB. Characterization of heat shock protein expression in apple peel under field and laboratory conditions[J]. J Am Soc Hortic Sci,2001,126(5):564−570. doi: 10.21273/JASHS.126.5.564
[38] 尹兵兵,潘凌云,付畅. 逆境下植物MAPK级联途径基因的表达调控[J]. 分子植物育种,2022,20(10):3257−3265. Yin BB,Pan LY,Fu C. Regulation of gene expression of plant MAPK cascade pathway under environmental[J]. Molecular Plant Breeding,2022,20(10):3257−3265.
[39] Huang X,Wang WL,Gong T,Wickell D,Kuo LY,et al. The flying spider-monkey tree fern genome provides insights into fern evolution and arborescence[J]. Nat Plants,2022,8(5):500−512. doi: 10.1038/s41477-022-01146-6
[40] Wanner LA,Li GQ,Ware D,Somssich IE,Davis KR. The phenylalanine ammonia-lyase gene family in Arabidopsis thaliana[J]. Plant Mol Biol,1995,27(2):327−338. doi: 10.1007/BF00020187
[41] Shirley BW,Kubasek WL,Storz G,Bruggemann E,Koornneef M,et al. Analysis of Arabidopsis mutants deficient in flavonoid biosynthesis[J]. Plant J,1995,8(5):659−671. doi: 10.1046/j.1365-313X.1995.08050659.x
[42] Ni R,Niu M,Fu J,Tan H,Zhu TT,et al. Molecular and structural characterization of a promiscuous chalcone synthase from the fern species Stenoloma chusanum[J]. J Integr Plant Biol,2022,64(10):1935−1951. doi: 10.1111/jipb.13335
[43] 林江波,王伟英,邹晖,戴艺民. 基于转录组测序的铁皮石斛黄酮代谢途径及相关基因解析[J]. 福建农业学报,2019,34(9):1019−1025. Lin JB,Wang WY,Zou H,Dai YM. Transcriptome analysis on pathway of and genes Related to flavonoid synthesis in Dendrobium officinale[J]. Fujian Journal of Agricultural Sciences,2019,34(9):1019−1025.
[44] Christensen AB,Gregersen PL,Schröder J,Collinge DB. A chalcone synthase with an unusual substrate preference is expressed in barley leaves in response to UV light and pathogen attack[J]. Plant Mol Biol,1998,37(5):849−857. doi: 10.1023/A:1006031822141
[45] Rudrapal M,Khan J,Dukhyil AAB,Alarousy RMII,Attah EI,et al. Chalcone scaffolds,bioprecursors of flavonoids:chemistry,bioactivities,and pharmacokinetics[J]. Molecules,2021,26(23):7177. doi: 10.3390/molecules26237177
[46] Van Tunen AJ,Mur LA,Recourt K,Gerats AG,Mol JN. Regulation and manipulation of flavonoid gene expression in anthers of petunia:the molecular basis of the Po mutation[J]. Plant Cell,1991,3(1):39−48.
[47] Yonekura-Sakakibara K,Higashi Y,Nakabayashi R. The origin and evolution of plant flavonoid metabolism[J]. Front Plant Sci,2019,10:943. doi: 10.3389/fpls.2019.00943
[48] Owens DK,Alerding AB,Crosby KC,Bandara AB,Westwood JH,Winkel BSJ. Functional analysis of a predicted flavonol synthase gene family in Arabidopsis[J]. Plant Physiol,2008,147(3):1046−1061. doi: 10.1104/pp.108.117457
[49] Sudheeran PK,Ovadia R,Galsarker O,Maoz I,Sela N,et al. Glycosylated flavonoids:fruit′s concealed antifungal arsenal[J]. New Phytol,2020,225(4):1788−1798. doi: 10.1111/nph.16251
[50] 陈雪菲,张映雪,黄贤燕,王全喜,曹建国,王幼芳. 蕨类植物花青素和原花青素成分及含量分析[J]. 植物科学学报,2020,38(6):820−830. doi: 10.11913/PSJ.2095-0837.2020.60820 Chen XF,Zhang YX,Huang XY,Wang QX,Cao JG,Wang YF. Composition and content analysis of anthocyanidins and proanthocyanidins in ferns[J]. Plant Science Journal,2020,38(6):820−830. doi: 10.11913/PSJ.2095-0837.2020.60820
[51] 肖珍. 黄酮醇和花青素在植物抗逆中的功能差异研究[D]. 济南: 山东大学, 2018: 1-20. [52] Taylor LP,Grotewold E. Flavonoids as developmental regulators[J]. Curr Opin Plant Biol,2005,8(3):317−323. doi: 10.1016/j.pbi.2005.03.005
-
其他相关附件
-
PDF格式
林泓 附件 点击下载(640KB)
-




 下载:
下载: