Genetic diversity and genetic structure analysis of wild Chinese Rosa roxburghii Tratt. germplasm resources
-
摘要:
基于10对EST-SSR引物,对中国8个省29个居群的261份野生刺梨(Rosa roxburghii Tratt.)进行毛细管电泳检测,探究其遗传多样性,并进行聚类分析。结果显示,共扩增出了95个等位基因,位点多态性信息含量(PIC)平均值为0.568;群体水平上,平均等位基因数(NA)和有效等位基因数(NE)分别为3.131和2.331,平均观测杂合度(HO)和预期杂合度(HE)分别为0.508和0.488,平均Shannon’s信息指数(I)为0.858,表明野生刺梨种质具有较高的遗传多样性水平。群体遗传分化分析结果显示,平均遗传分化系数(FST)为0.067,基因流(Nm)为4.511,说明刺梨群体间基因流动较频繁。分子方差分析(AMOVA)结果表明刺梨的遗传变异主要来源于居群内(93.64%)。各居群间的Nei氏标准遗传距离范围为0.054~1.269,平均值为0.657,与地理距离表现为显著相关(r=0.467,P<0.000 1)。聚类分析和PCA结果均将29个居群分为3个分支。研究结果说明中国野生刺梨资源核心分布区在我国西南地区。
Abstract:This study explored the genetic diversity and structure of 261 wild Rosa roxburghii Tratt. samples from 29 populations across eight provinces in China, utilizing 10 pairs of polymorphic EST-SSR primers and capillary electrophoresis, providing a clear genetic background for the collection and breeding of R. roxburghii germplasm resources. Results identified 95 alleles among the 261 germplasm materials. The average polymorphism information content of the loci was 0.568. At the population level, the average number of alleles and effective number of alleles were 3.131 and 2.331, respectively, with observed heterozygosity and expected heterozygosity averaging 0.508 and 0.488. The average Shannon’s information index was 0.858, indicating substantial genetic diversity, likely due to the extensive range of sampling sites. Analysis of population genetic differentiation revealed an average genetic differentiation coefficient of 0.067 and gene flow of 4.511, suggesting considerable inter-population gene flow. Analysis of molecular variance (AMOVA) indicated that most genetic variation in R. roxburghii came from within populations (r=0.467, P<0.000 1). Nei’s standard genetic distance among populations ranged from 0.054 to 1.269, with an average of 0.657, and was significantly related to geographical distance. Clustering analysis grouped the 29 populations into three main clusters, which was related to geographical location. These results highlight southwestern China as a core distribution area for wild R. roxburghii resources, providing theoretical guidance for the formulation of R. roxburghii conservation strategies.
-
Keywords:
- Rosa roxburghii /
- SSR primers /
- Genetic diversity /
- Genetic structure
-
刺梨(Rosa roxburghii Tratt.),又名缫丝花,蔷薇科蔷薇属(Rosa)植物,多为丛生,多年生落叶灌木,二倍体植物,属于具有一定自花结实能力的异花授粉植物[1]。刺梨是我国特有的野生植物,主要分布在贵州、湖南、江西、云南、湖北、重庆、陕西、四川等地[2]。刺梨是我国新兴的第三代水果,果实中VC含量居各类水果之冠,具有很高的药用价值和保健功能。我国野生刺梨资源丰富,但刺梨遗传改良成效不明显,鲜有对中国野生刺梨资源的遗传多样性评价和种质资源保存的研究。因此,全面了解中国野生刺梨遗传多样性,掌握其遗传背景,对刺梨资源收集保存、重要优异基因挖掘以及遗传改良具有重要意义。
用于研究遗传多样性的标记方法不断更新,最早是利用形态、同工酶等传统的方法[3, 4]。随着生物技术的发展,AFLP、RAPD、SSR、ISSR、SNP等分子标记方法也在植物中得到了很好的应用[5, 6]。其中,EST-SSR多态性丰富、稳定性强、操作简单,是评价遗传多样性最有效的方法之一[7, 8],已被广泛应用于农作物、果树、林木的遗传分析[9-12]。在以往刺梨的遗传多样性研究中,主要集中于贵州省内的种质,有少数研究收集了除贵州省以外的种质,但有的居群仅有一个样本,代表性欠缺。
本研究以来源于全国野生刺梨资源丰富的8个省份的29个居群为研究材料,利用刺梨转录组和近缘物种EST序列开发的SSR标记,全面探究野生刺梨的遗传多样性和遗传结构,旨在了解中国野生刺梨遗传背景,为刺梨种质资源的核心种质库构建、种质创新和利用以及优良基因挖掘奠定基础。
1. 材料与方法
1.1 实验材料
本研究所需材料均来自贵州省黔南州刺梨种质资源库,该资源库在2020−2021年收集了我国野生刺梨主要分布区的8个省份的种质,建库时根据自然群体大小进行采样;在同一地区采样时为了避免重复,每株距离30 m以上,于贵州省黔南洲林业局温室进行扩繁,于2022年移栽在都匀市马鞍山林场,小区规模为4~10株,株行距为3 m×2 m 。该林场海拔约900 m,年平均气温16.1 ℃,年平均降水量1 431.1 mm,无霜期300 d左右。
2022年9-11月采集新鲜无病虫害刺梨叶片,根据现有的刺梨种质,共采用261份,来自29个居群(表1),于 −80 ℃冰箱保存备用。
表 1 野生刺梨采样数量情况Table 1. Number of wild Rosa roxburghii samples编号
Code居群
Population数量
Number编号
Code居群
Population数量
NumberDS 贵州省独山县 12 PL 陕西省平利县 3 DY 贵州省都匀市 8 PY 江西省鄱阳县 5 FQ 贵州省福泉市 6 BJ 湖南省保靖县 5 GD 贵州省贵定县 36 ES 湖北省恩施市 8 HS 贵州省惠水县 3 XE 湖北省宣恩县 10 LL 贵州省龙里县 3 DJY 四川省都江堰 3 PT 贵州省平塘县 4 MN 四川省冕宁县 12 SD 贵州省三都县 15 AZ 四川省安州区 5 WA 贵州省瓮安县 20 YY 重庆市酉阳县 13 XR 贵州省兴仁市 4 BN 重庆市巴南区 16 LZ 贵州省六枝特区 5 LPQ 重庆市梁平区 4 SC 贵州省水城区 6 ZX 云南省镇雄市 10 CS 贵州省长顺县 6 YJ 云南省盐津县 17 LP 贵州省黎平县 4 SF 云南省水富市 6 NQ 陕西省宁强县 12 1.2 实验方法
1.2.1 DNA提取与检测
选取刺梨新鲜叶片30~50 mg,利用CTAB法提取DNA[13],并将DNA的浓度统一稀释至3~5 ng/μL以备扩增使用。
1.2.2 SSR引物来源及合成
刺梨SSR引物一部分来自刺梨前期转录组的SSR引物[14],另一部分由其近缘物种萨曼莎月季(R. chinensis Jacq.)的EST序列开发获得。引物设计参考鄢秀芹[15]的研究。
1.2.3 引物筛选
从备选引物中挑选多态性引物,利用地理位置较远的刺梨样本检测SSR位点多态性。PCR反应体系和程序同Lu等[16]的方法。
毛细管电泳检测中,共使用3条TP-M13-SSR引物,分别为在正向引物的5'端加M13尾端( TGTAAAACGACGGCCAGT)、普通的反向引物以及带有 ROX(red)、HEX(green)和 FAM(blue)3 种M13荧光标记的引物。PCR扩增体系为:DNA模板1.4 μL (5 ng/μL),荧光引物1 μL,2 × Taq Mix 6 μL,反向引物1 μL,正向引物0.5 μL,ddH2O 6 μL。PCR反应程序为:95 ℃预变性5 min;95 ℃变性40 s,55 ℃退火45 s,72 ℃ 延伸40 s,共25个循环;第2轮程序为:95 ℃变性 40 s,53 ℃退火 45 s,72 ℃延伸 40 s,共10个循环;72 ℃延伸10 min。
1.2.4 基因分型
将带不同荧光的PCR扩增产物混合在一起,使用ABI 3730XL DNA测序仪进行毛细管电泳检测。使用GeneMarker v2.2.0软件对毛细管电泳结果进行分析,将SSR分型结果录入Excel 2003软件中。毛细管电泳检测委托生工生物工程股份有限公司进行。
1.2.5 数据分析
采用GenAIEx 6.5软件估算不同SSR位点及各群体的遗传多样性参数,包括:等位基因数目(Number of alleles,NA)、有效等位基因数目(Effective number of alleles,NE)、观测杂合度(Observed heterozygosity,Ho)、期望杂合度(Effective heterozygosity,HE)、Shannon’s信息指数(Shannon information index,I)、群体遗传分化系数(Within-population differentiation coefficient,FST)、总近交系数(Total inbreeding coefficient, FIS)、基因流(Gene flow,Nm),Nei氏标准遗传距离(Nei’s standard genetic distance),并对刺梨群体进行PCA分析。采用Cervus 3.0软件计算位点多态性信息含量(Polymorphism information content, PIC)。
利用GenAIEx 6.5软件进行分子方差分析(AMOVA),估算居群间、居群内遗传方差比例。结合成对居群的遗传距离,利用GenAIEx 6.5软件进行Mantel相关性检验,用R语言做相关性图;最后基于Nei氏标准遗传距离,用NTsyspc 2.1软件基于UPGMA算法进行聚类分析。
2. 结果与分析
2.1 多态性引物筛选
本研究对60对引物进行初筛,其中20对来自刺梨转录组,40对来自萨曼莎月季的EST序列。选取地理距离较远的8份材料对60对引物进行筛选,经检测,38对引物能扩增出清晰可见的条带。
对38对引物进行毛细管电泳检测,发现引物Sam1在种质重庆西南区2号、湖南保靖14号中的毛细管电泳结果良好(图1),该引物在不同种质间的扩增片段大小均不相同,表明Sam1引物具有较好的多态性,能用于刺梨遗传多样性研究。经PCR扩增,毛细管电泳检测后,共筛选出10对多态性较高的引物(表2)。
表 2 刺梨多态性引物Table 2. Polymorphic primers of Rosa roxburghii引物
Primer引物序列(5′→3′)
Primer sequence(5′→3′)来源
Source重复基元
Repeated motif产物大小
Product size / bpRox1 F: AATATACACGAACAACAACCATCG
R: GGAACATGACCCCTTTTCTTATT刺梨
R. roxburghii(CTACA)4 147 Rox2 F: GAGTCATGTTGAATGATATTGGC
R: TTTCCTCTTTTCTTCTTTTTCCC刺梨 (TGGA)13 131 Rox3 F: TGAGAACCAAAAGAAACGACTACA
R: GTCACAAATTCACGAGCCATATT刺梨 (AT)8 146 Rox4 F: GAGAGAGTAGCCTTTGATGAGGA
R: GAATGTCGTCAGGGAGCAGTAG刺梨 (CTC)7 146 Sam1 F: ATGCCCTTTTGATGTGCTGGTAGAG
R: TCGGATCTTCTTGTGCTGTTTCTCC月季
R. chinensis(AG)5 233 Sam2 F: TGGTGGCAAACAGGAGAAGATATGG
R: GCAACATCTGCACAACTGGAAGC月季 (GA)5 211 Sam3 F: GCAACAACAGCAACAACTTCCTTTG
R: GTTGGAGTGGCAGTTGATGCTTATG月季 (CAA)5 245 Sam4 F: GTTGCTCCACAGATGCCTCAGTC
R: TGGTTGAAATTGCTGCTGTTGTTGG月季 (CAG)5 258 Sam5 F: GAACCAAGTCCTTCCGCAAA
R: CCAACATAGCGGTTGCTTGA月季 (CTT)5 223 Sam6 F: GTGTACTATCGAGCCGATCCAGGAG
R: AACCACGCTTCAATGGCTCATCC月季 (CT)5 200 2.2 SSR位点的遗传多样性参数
利用GenAIEx 6.5软件估算出10对EST-SSR引物在261份野生刺梨材料的遗传多样性指数(表3),结果显示,共检测出95个多态性基因位点,每对引物的等位基因数为5~14,平均为9.5个,NE平均值为2.311;引物的PIC值在0.227~0.782范围波动,平均值为0.568,其中有9对引物的PIC值都大于0.5,为高度多态性信息引物。平均观测杂合度和预期杂合度分别为0.508和0.488;I平均值为0.858,因此,筛选确定的10对EST-SSR引物表现出高度多态性,在供试的261份刺梨种质中反映出丰富的遗传多样性。
表 3 10对SSR引物在261份刺梨种质中的遗传信息Table 3. Genetic information of 10 SSR primers in 261 Rosa roxburghii germplasms引物Primer 等位基因数 Number of alleles 有效等位基因数 Effective number of alleles Shannon’s指数Shannon information index 观测杂合度Observed heterozygosity 期望杂合度Effective heterozygosity 多态信息含量Polymorphism information content Sam1 7 1.305 0.307 0.073 0.180 0.227 Sam2 13 1.640 0.476 0.060 0.288 0.503 Sam3 9 1.832 0.661 0.394 0.401 0.456 Sam4 8 2.365 0.986 0.731 0.551 0.548 Sam5 11 2.284 0.913 0.708 0.550 0.576 Sam6 13 2.078 0.813 0.538 0.481 0.521 Rox1 14 2.688 0.997 0.457 0.550 0.750 Rox2 5 2.410 0.903 0.714 0.554 0.599 Rox3 11 3.597 1.323 0.758 0.690 0.782 Rox4 11 3.115 1.200 0.649 0.634 0.718 平均值 10.2 2.311 0.858 0.508 0.488 0.568 2.3 刺梨群体遗传多样性
利用GenAlex 6.5软件估算来自不同居群的刺梨的遗传多样性(表4),发现各居群的NA波动范围为1.600~4.000,平均值为3.131;NE为1.500~3.223,平均值为2.331;I为0.381~1.233,平均值为0.858;Ho和HE都是陕西省宁强县(NQ)的最大(0.731和0.645),贵州省黎平县(LP)的观测杂合度和期望杂合度最低(0.300和0.482),且Ho均大于HE。
表 4 不同居群平均多样性指数Table 4. Average diversity index of different populations群体
Population等位基因数
Number of
alleles有效等位基因数
Effective number
of allelesShannon’s指数
Shannon information
index观测杂合度
Observed
heterozygosity期望杂合度
Observed
heterozygosity固定指数
Fixation index多态性位点比率
Polymorphic
site rate / %CS 3.300 2.170 0.868 0.322 0.479 0.346 90.00 DS 3.700 2.780 1.035 0.656 0.572 −0.075 100.00 DY 3.400 2.452 0.941 0.489 0.530 0.064 100.00 FQ 2.900 2.320 0.852 0.463 0.504 0.080 100.00 GD 5.400 2.433 1.013 0.545 0.523 −0.013 100.00 HS 2.000 1.678 0.584 0.450 0.376 −0.192 80.00 LP 2.600 2.078 0.786 0.300 0.482 0.390 100.00 LL 2.600 2.161 0.763 0.517 0.449 −0.186 80.00 PT 2.300 1.877 0.672 0.417 0.426 0.002 90.00 SD 3.500 2.563 0.959 0.661 0.537 −0.165 100.00 WA 3.900 2.536 0.987 0.587 0.535 0.035 100.00 XR 2.300 2.032 0.666 0.600 0.419 −0.472 80.00 LZ 2.400 2.117 0.731 0.675 0.435 −0.561 70.00 SC 2.500 2.008 0.666 0.583 0.391 −0.503 70.00 ES 3.500 2.585 1.033 0.419 0.588 0.307 100.00 XE 3.700 2.533 0.997 0.401 0.540 0.272 100.00 BJ 3.500 2.952 1.062 0.647 0.588 −0.076 90.00 PY 2.500 2.121 0.705 0.340 0.412 0.138 70.00 NQ 4.600 3.223 1.233 0.731 0.645 −0.118 100.00 PL 1.800 1.530 0.476 0.433 0.306 −0.363 70.00 DJY 1.600 1.500 0.381 0.300 0.238 −0.300 40.00 MN 3.500 2.494 0.953 0.553 0.522 0.003 90.00 AZ 3.100 2.489 0.898 0.360 0.503 0.254 90.00 SF 3.000 2.154 0.848 0.440 0.495 0.101 90.00 YJ 4.000 2.877 1.076 0.490 0.562 0.182 100.00 ZX 3.000 2.382 0.894 0.411 0.522 0.286 100.00 LPQ 2.500 2.160 0.699 0.592 0.418 −0.446 80.00 YY 3.700 2.769 1.059 0.680 0.587 −0.185 100.00 BN 4.000 2.631 1.044 0.673 0.565 −0.067 100.00 平均值 3.131 2.331 0.858 0.508 0.488 −0.019 88.97 2.4 刺梨居群遗传分化
本研究发现,29个刺梨居群内FST值为0.035~0.042,平均值为0.067,属低等遗传分化程度,居群间基因流值波动较大,为1.516~6.805,平均值为4.511,群体间基因流动较大。29个刺梨居群在各位点上平均FIS为0.276,其中在Sam4、Sam5位点上为负值(表5)。
表 5 各SSR位点F-statistics及基因流Table 5. F-statistics and gene flow for each SSR microsatellite locus位点Locus 居群内分化系数
Within-population differentiation coefficient总近交系数
Total inbreeding coefficient基因流Gene flow Sam1 0.142 0.819 1.516 Sam2 0.116 0.918 1.899 Sam3 0.041 0.110 5.828 Sam4 0.039 −0.306 6.084 Sam5 0.091 −0.170 2.503 Sam6 0.044 0.242 5.400 Rox1 0.084 0.623 2.716 Rox2 0.040 0.268 6.008 Rox3 0.038 0.195 6.352 Rox4 0.035 0.065 6.805 平均 值 0.067 0.276 4.511 刺梨居群内变异分量占总变异的93.64%,居群间遗传变异占总变异的6.36%(表6),居群间和居群内遗传变异差异显著,说明群体间存在显著的遗传结构,且刺梨的遗传变异主要是由居群内变异引起的。
表 6 刺梨29个居群分子方差分析Table 6. Analysis of molecular variance (AMOVA)of 29 Rosa roxburghii populations变异来源
Variance source自由度
df离均差平方和
SS均方
MS估计方差
Estimated variance变异百分比
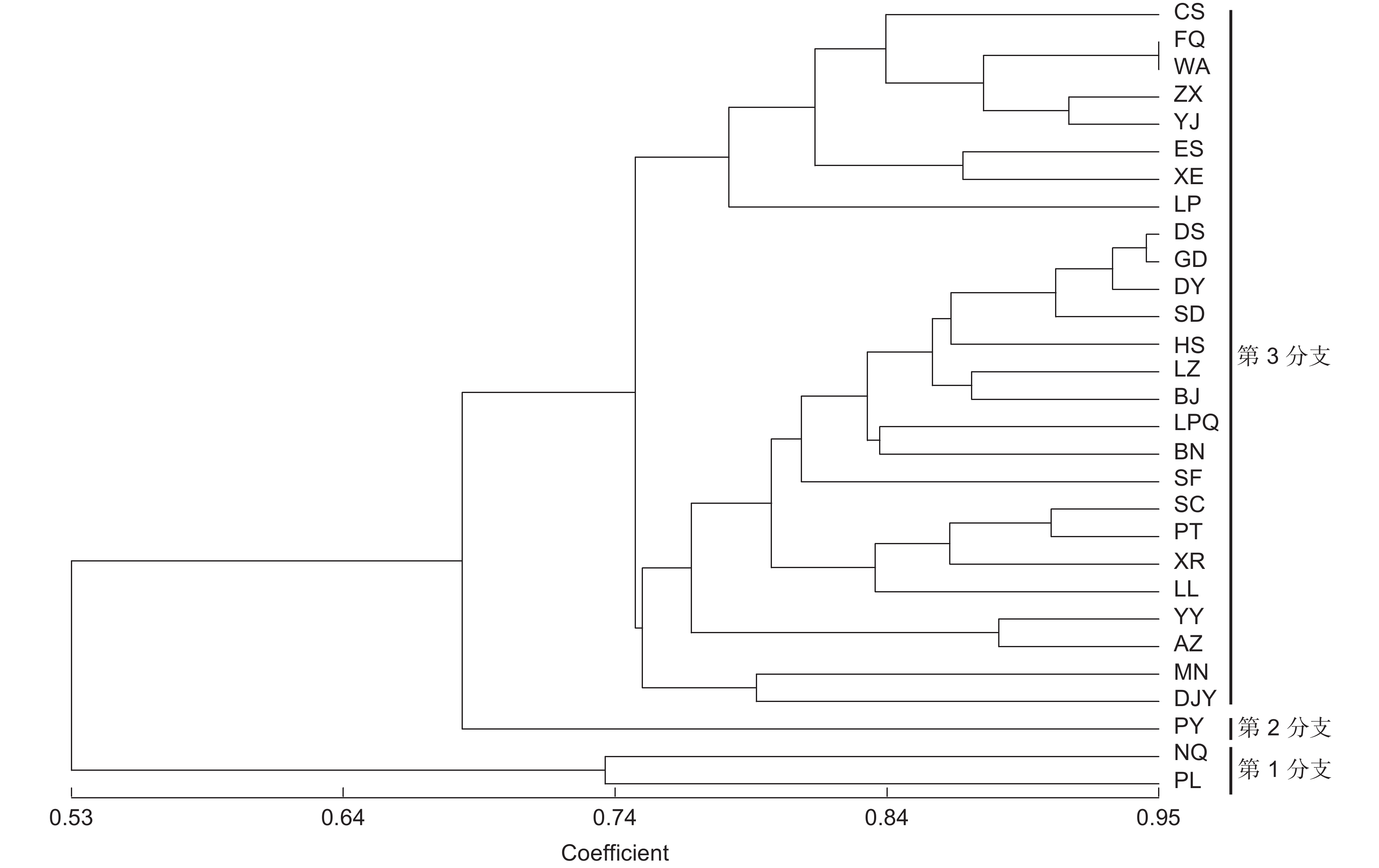
Percentage variation / %群体间 28 213.437 7.623 0.211 6.36 群体内 493 1 509.647 6.218 3.109 93.64 总计 521 1 723.084 3.320 100 经GenAlex软件分析得知,Nei氏遗传一致度范围在0.281~0.948;遗传距离与遗传一致性呈反比,范围很广,在0.054~1.269,平均值为0.657。在遗传相似系数为0.70时,将29个居群分为3个分支,第1分支包括2个居群,分别来自陕西省的平利县(PL)和陕西省宁强县(NQ);第2分支仅有江西鄱阳(PY)1个居群,其他的26个居群组成第3分支(图2)。
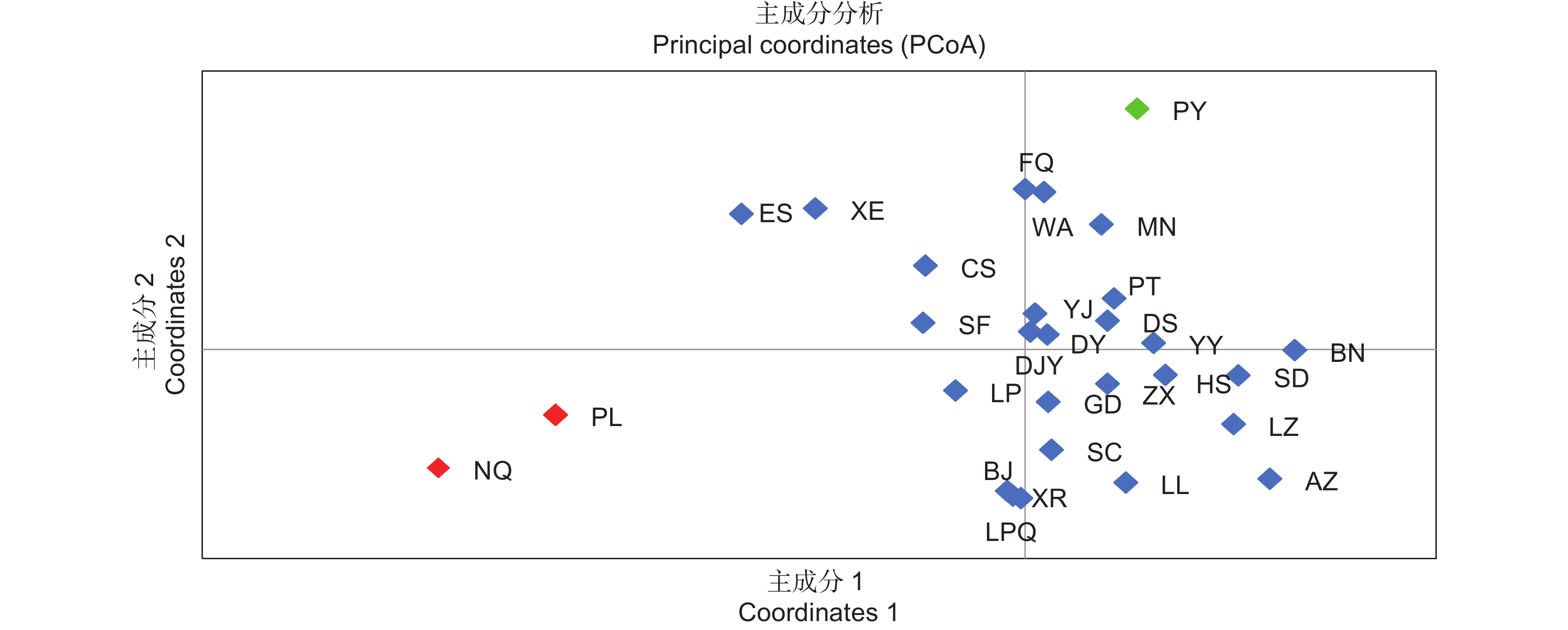
按照主成分1贡献值为33.97%,主成分2贡献值为20.57%,把29个群体分成3个分支(图3),分支情况与聚类结果一致。
根据两两居群间的地理距离,结合Nei氏标准遗传距离,进行Mantel相关性检验(r=0.467,P<0.000 1,图4),表明居群间的Nei氏标准遗传距离与地理距离有显著的相关性,地理阻碍是刺梨群体遗传分化的重要因素。
3. 讨论
3.1 刺梨遗传多样性分析
研究发现,遗传多样性越高,即遗传变异越丰富,植物对环境变化的适应能力就越强。本研究采集的样本范围广泛,是目前最具代表性的种质资源。本研究基于SSR分子标记对261份刺梨种质资源遗传多样性进行分析,结果表明,平均NE、I、HE分别为2.331、0.858、0.488,与张怀山等[14]的研究结果相近(NE = 2.474、I = 0.920、HE = 0.585);高于潘凤等[17]的研究(NE = 1.618、I = 0.534、HE = 0.358),这是因为采样范围和方式不同。本研究涉及到了8个省份,且每个居群至少3个样本,范围更加广泛,代表性更强。与同样基于SSR标记的其他蔷薇科植物比较,相近于山樱花(Prunus serrulata Lindl.)(I = 0.939、HE = 0.488)[18],略低于野生的云南多依(Docynia delavayi Schneid.)(I = 0.884、HE = 0.522)[19]、低于康定樱桃(Cerasus tatsienensis Batal.)(I = 0.935、HE =0.529)[20],高于现代月季(I = 0.421、HE = 0.277)[21]、黄毛草莓(Fragaria nilgerrensis L.)(I = 0.423、HE = 0.275)[22],说明野生刺梨群体遗传多样性整体属于中等偏上的水平,这与刺梨倾向于异交的繁育系统相关。以往研究表明,刺梨存在自交不亲和的现象[1],本研究发现野生刺梨的29个居群中,平均固定指数(FIS)为-0.019,说明总体观测杂合度高于期望杂合度,表明刺梨各居群中存在杂合子剩余的情况,显示了刺梨异交的生殖特性。
3.2 遗传分化分析
低的遗传分化是由于高的基因流引起,往往栖息地生境连通性好会导致基因流动水平高。本研究发现,刺梨物种水平上Nm为4.511,这与低水平的遗传分化相匹配(FST=0.067),低于李楠玉等[23]的野生刺梨FST值(0.143)。与其他中国野生植物相比,刺梨的遗传分化系数低于长柄双花木(Disanthus cercidifolius Maxim.,0.403) [24]、伯乐树(Bretschneidera sinensis Hemsl.,0.214)[25]、云南依(0.311)[26]和半夏(Pinellia ternata Breit.,0.909)[27],表明刺梨的遗传分化水平较低,各居群间基因流频繁,这可能由以下原因导致:一是刺梨是虫媒、风媒植物,这些授粉方式一般基因流较大,使得花粉交流频繁[1];二是由于鸟、兽觅食野生刺梨的种子,加上各地种子由人为传播携带进行频繁地交换[28]。另外,部分刺梨材料来自贵州、四川、云南、重庆等西南地区的河沟旁,河边的刺梨种子会顺着河道扩散至地势较低的地方,这些因素都导致种子流频繁[29]。分子方差分析结果显示,刺梨的遗传变异主要来源于居群内(93.64%),而居群之间的变异较小(6.36%),这与前人的研究结果一致[14, 23]。
本研究结果表明,刺梨居群地理距离与遗传距离之间存在正相关,提示刺梨的野生居群间存在地理隔离,说明刺梨野生居群在通过虫媒、风媒传播花粉时,由于贵州、云南等西南地区的高山阻碍和刺梨个体花期不重叠导致花粉流存在限制,且由于动物或者人为引起的种子流都更容易发生在近距离范围内,使刺梨出现片段化分布[30]。聚类分析和PCA分析的结果一致,将29个居群分为3大分支。其中陕西省的2个居群NQ和PL归为1个分支,江西省的PY单独为1个分支,第3分支均属西南地区的种质,此结果与地理位置有关,也可说明中国西南地区为刺梨的核心分布区,例如第3分支中湖北的ES和XE,四川的DJY、AZ和MN也证实了聚类结果与地理位置相关。
4. 结论
本研究对来自中国8个省29个居群的261份野生刺梨种质进行遗传多样性分析,结果表明中国野生刺梨遗传多样性较高,具有较高的进化潜力。刺梨整体遗传分化低,这是由于居群间高的基因流及异交繁育方式而导致。遗传距离与地理距离显著相关。居群聚类结果把29个居群分为3个分支,最大的第3分支来自于我国西南,推测该区域为刺梨核心分布区。
-
表 1 野生刺梨采样数量情况
Table 1 Number of wild Rosa roxburghii samples
编号
Code居群
Population数量
Number编号
Code居群
Population数量
NumberDS 贵州省独山县 12 PL 陕西省平利县 3 DY 贵州省都匀市 8 PY 江西省鄱阳县 5 FQ 贵州省福泉市 6 BJ 湖南省保靖县 5 GD 贵州省贵定县 36 ES 湖北省恩施市 8 HS 贵州省惠水县 3 XE 湖北省宣恩县 10 LL 贵州省龙里县 3 DJY 四川省都江堰 3 PT 贵州省平塘县 4 MN 四川省冕宁县 12 SD 贵州省三都县 15 AZ 四川省安州区 5 WA 贵州省瓮安县 20 YY 重庆市酉阳县 13 XR 贵州省兴仁市 4 BN 重庆市巴南区 16 LZ 贵州省六枝特区 5 LPQ 重庆市梁平区 4 SC 贵州省水城区 6 ZX 云南省镇雄市 10 CS 贵州省长顺县 6 YJ 云南省盐津县 17 LP 贵州省黎平县 4 SF 云南省水富市 6 NQ 陕西省宁强县 12 表 2 刺梨多态性引物
Table 2 Polymorphic primers of Rosa roxburghii
引物
Primer引物序列(5′→3′)
Primer sequence(5′→3′)来源
Source重复基元
Repeated motif产物大小
Product size / bpRox1 F: AATATACACGAACAACAACCATCG
R: GGAACATGACCCCTTTTCTTATT刺梨
R. roxburghii(CTACA)4 147 Rox2 F: GAGTCATGTTGAATGATATTGGC
R: TTTCCTCTTTTCTTCTTTTTCCC刺梨 (TGGA)13 131 Rox3 F: TGAGAACCAAAAGAAACGACTACA
R: GTCACAAATTCACGAGCCATATT刺梨 (AT)8 146 Rox4 F: GAGAGAGTAGCCTTTGATGAGGA
R: GAATGTCGTCAGGGAGCAGTAG刺梨 (CTC)7 146 Sam1 F: ATGCCCTTTTGATGTGCTGGTAGAG
R: TCGGATCTTCTTGTGCTGTTTCTCC月季
R. chinensis(AG)5 233 Sam2 F: TGGTGGCAAACAGGAGAAGATATGG
R: GCAACATCTGCACAACTGGAAGC月季 (GA)5 211 Sam3 F: GCAACAACAGCAACAACTTCCTTTG
R: GTTGGAGTGGCAGTTGATGCTTATG月季 (CAA)5 245 Sam4 F: GTTGCTCCACAGATGCCTCAGTC
R: TGGTTGAAATTGCTGCTGTTGTTGG月季 (CAG)5 258 Sam5 F: GAACCAAGTCCTTCCGCAAA
R: CCAACATAGCGGTTGCTTGA月季 (CTT)5 223 Sam6 F: GTGTACTATCGAGCCGATCCAGGAG
R: AACCACGCTTCAATGGCTCATCC月季 (CT)5 200 表 3 10对SSR引物在261份刺梨种质中的遗传信息
Table 3 Genetic information of 10 SSR primers in 261 Rosa roxburghii germplasms
引物Primer 等位基因数 Number of alleles 有效等位基因数 Effective number of alleles Shannon’s指数Shannon information index 观测杂合度Observed heterozygosity 期望杂合度Effective heterozygosity 多态信息含量Polymorphism information content Sam1 7 1.305 0.307 0.073 0.180 0.227 Sam2 13 1.640 0.476 0.060 0.288 0.503 Sam3 9 1.832 0.661 0.394 0.401 0.456 Sam4 8 2.365 0.986 0.731 0.551 0.548 Sam5 11 2.284 0.913 0.708 0.550 0.576 Sam6 13 2.078 0.813 0.538 0.481 0.521 Rox1 14 2.688 0.997 0.457 0.550 0.750 Rox2 5 2.410 0.903 0.714 0.554 0.599 Rox3 11 3.597 1.323 0.758 0.690 0.782 Rox4 11 3.115 1.200 0.649 0.634 0.718 平均值 10.2 2.311 0.858 0.508 0.488 0.568 表 4 不同居群平均多样性指数
Table 4 Average diversity index of different populations
群体
Population等位基因数
Number of
alleles有效等位基因数
Effective number
of allelesShannon’s指数
Shannon information
index观测杂合度
Observed
heterozygosity期望杂合度
Observed
heterozygosity固定指数
Fixation index多态性位点比率
Polymorphic
site rate / %CS 3.300 2.170 0.868 0.322 0.479 0.346 90.00 DS 3.700 2.780 1.035 0.656 0.572 −0.075 100.00 DY 3.400 2.452 0.941 0.489 0.530 0.064 100.00 FQ 2.900 2.320 0.852 0.463 0.504 0.080 100.00 GD 5.400 2.433 1.013 0.545 0.523 −0.013 100.00 HS 2.000 1.678 0.584 0.450 0.376 −0.192 80.00 LP 2.600 2.078 0.786 0.300 0.482 0.390 100.00 LL 2.600 2.161 0.763 0.517 0.449 −0.186 80.00 PT 2.300 1.877 0.672 0.417 0.426 0.002 90.00 SD 3.500 2.563 0.959 0.661 0.537 −0.165 100.00 WA 3.900 2.536 0.987 0.587 0.535 0.035 100.00 XR 2.300 2.032 0.666 0.600 0.419 −0.472 80.00 LZ 2.400 2.117 0.731 0.675 0.435 −0.561 70.00 SC 2.500 2.008 0.666 0.583 0.391 −0.503 70.00 ES 3.500 2.585 1.033 0.419 0.588 0.307 100.00 XE 3.700 2.533 0.997 0.401 0.540 0.272 100.00 BJ 3.500 2.952 1.062 0.647 0.588 −0.076 90.00 PY 2.500 2.121 0.705 0.340 0.412 0.138 70.00 NQ 4.600 3.223 1.233 0.731 0.645 −0.118 100.00 PL 1.800 1.530 0.476 0.433 0.306 −0.363 70.00 DJY 1.600 1.500 0.381 0.300 0.238 −0.300 40.00 MN 3.500 2.494 0.953 0.553 0.522 0.003 90.00 AZ 3.100 2.489 0.898 0.360 0.503 0.254 90.00 SF 3.000 2.154 0.848 0.440 0.495 0.101 90.00 YJ 4.000 2.877 1.076 0.490 0.562 0.182 100.00 ZX 3.000 2.382 0.894 0.411 0.522 0.286 100.00 LPQ 2.500 2.160 0.699 0.592 0.418 −0.446 80.00 YY 3.700 2.769 1.059 0.680 0.587 −0.185 100.00 BN 4.000 2.631 1.044 0.673 0.565 −0.067 100.00 平均值 3.131 2.331 0.858 0.508 0.488 −0.019 88.97 表 5 各SSR位点F-statistics及基因流
Table 5 F-statistics and gene flow for each SSR microsatellite locus
位点Locus 居群内分化系数
Within-population differentiation coefficient总近交系数
Total inbreeding coefficient基因流Gene flow Sam1 0.142 0.819 1.516 Sam2 0.116 0.918 1.899 Sam3 0.041 0.110 5.828 Sam4 0.039 −0.306 6.084 Sam5 0.091 −0.170 2.503 Sam6 0.044 0.242 5.400 Rox1 0.084 0.623 2.716 Rox2 0.040 0.268 6.008 Rox3 0.038 0.195 6.352 Rox4 0.035 0.065 6.805 平均 值 0.067 0.276 4.511 表 6 刺梨29个居群分子方差分析
Table 6 Analysis of molecular variance (AMOVA)of 29 Rosa roxburghii populations
变异来源
Variance source自由度
df离均差平方和
SS均方
MS估计方差
Estimated variance变异百分比
Percentage variation / %群体间 28 213.437 7.623 0.211 6.36 群体内 493 1 509.647 6.218 3.109 93.64 总计 521 1 723.084 3.320 100 -
[1] 陈明,陈继平. 刺梨授粉结实习性研究[J]. 绵阳农专学报,1993,10(3-4):69−70. Chen M,Chen JP. A study of habit of pollination and fruitage on Roxburgh rose[J]. Journal of Mianyang Agricultural College,1993,10(3-4):69−70.
[2] 胡红菊. 我国野生刺梨资源开发利用探讨[J]. 中国果树,2009(3):71−72. [3] 徐东旭,姜翠棉,宗绪晓. 蚕豆种质资源形态标记遗传多样性分析[J]. 植物遗传资源学报,2010,11(4):399−406. Xu DX,Jiang CM,Zong XX. Phenotypic diversity of faba bean (Vicia faba L.) germplasm resources[J]. Journal of Plant Genetic Resources,2010,11(4):399−406.
[4] Petit RJ,El Mousadik A,Pons O. Identifying populations for conservation on the basis of genetic markers[J]. Conser Biol,1998,12(4):844−855.
[5] Powell W,Morgante M,Andre C,Hanafey M,Vogel J,et al. The comparison of RFLP,RAPD,AFLP and SSR (microsatellite) markers for germplasm analysis[J]. Mol Breed,1996,2(3):225−238.
[6] Tang WJ,Wu TT,Ye J,Sun J,Jiang Y,et al. Erratum to:SNP-based analysis of genetic diversity reveals important alleles associated with seed size in rice[J]. BMC Plant Biol,2016,16(1):128.
[7] Tautz D. Hypervariability of simple sequences as a general source for polymorphic DNA markers[J]. Nucleic Acids Res,1989,17(16):6463−6471.
[8] Powell W,Machray GC,Provan J. Polymorphism revealed by simple sequence repeats[J]. Trends Plant Sci,1996,1(7):215−222.
[9] Liu G,Xie YJ,Zhang DQ,Chen HP. Analysis of SSR loci and development of SSR primers in Eucalyptus[J]. J Forest Res,2018,29(2):273−282.
[10] Singh AK,Chaurasia S,Kumar S,Singh R,Kumari J,et al. Identification,analysis and development of salt responsive candidate gene based SSR markers in wheat[J]. BMC Plant Biol,2018,18(1):249.
[11] Kaur S,Pembleton LW,Cogan NO,Savin KW,Leonforte T,et al. Transcriptome sequencing of field pea and faba bean for discovery and validation of SSR genetic markers[J]. BMC Genomics,2012,13(1):104.
[12] 孙浩男,李蓉,李明阳,郑妍,刘志杰,等. 栽培金鸡菊EST-SSR分子标记开发与亲缘关系分析[J]. 草地学报,2022,30(6):1430−1440. Sun HN,Li R,Li MY,Zheng Y,Liu ZJ,et al. Development of EST-SSR markers based on transcriptome and analysis of genetic relationship of cultivated coreopsis[J]. Acta Agrestia Sinica,2022,30(6):1430−1440.
[13] Porebski S,Bailey LG,Baum BR. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components[J]. Plant Mol Biol Rep,1997,15(1):8−15.
[14] 张怀山,鄢秀芹,鲁敏,王道平,安华明. 基于EST-SSR标记的贵州野生刺梨居群遗传多样性分析[J]. 中国农业科学,2017,50(6):1098−1108. Zhang HS,Yan XQ,Lu M,Wang DP,An HM. Analysis of the genetic diversity of wild Rosa roxburghii populations in Guizhou province based on EST-SSR marker[J]. Scientia Agricultura Sinica,2017,50(6):1098−1108.
[15] 鄢秀芹. 刺梨果实转录组分析及EST-SSR开发与应用[D]. 贵阳:贵州大学,2015:13−29. [16] Lu M,An HM,Li LL. Genome survey sequencing for the characterization of the genetic background of Rosa roxburghii Tratt and leaf ascorbate metabolism genes[J]. PLoS One,2016,11(2):e0147530.
[17] 潘凤. 刺梨的遗传多样性及谱系地理学研究[D]. 贵阳:贵州大学,2022:12−13. [18] 伊贤贵,陈洁,尤禄祥,从睿,王华辰,等. 山樱花群体遗传多样性的SSR分析[J]. 南京林业大学学报(自然科学版),2018,42(5):25−31. Yi XG,Chen J,You LX,Cong R,Wang HC,et al. Genetic diversity of Cerasus serrulata populations assessed by SSR markers[J]. Journal of Nanjing Forestry University (Natural Sciences Edition),2018,42(5):25−31.
[19] Peng JY,Shi C,Wang DW,Li SZ,Zhao XL,et al. Genetic diversity and population structure of the medicinal plant Docynia delavayi (Franch.) Schneid revealed by transcriptome based SSR markers[J]. J Appl Res Med Aroma Plants,2021,21:100294.
[20] 从睿. 康定樱桃居群遗传多样性的SSR分析[D]. 南京:南京林业大学,2018:19−28. [21] 吴高琼. 基于SSR分子标记的中国古老月季野生亲本分析[D]. 昆明:云南大学,2019:25−27. [22] 刁霞. 黄毛草莓(Fragarian nilgerrensis Schldl)EST-SSR引物开发及遗传多样性研究[D]. 昆明:云南大学,2019:25−31. [23] 李楠玉,张怀山,安华明,鲁敏. 基于ITS序列对刺梨遗传多样性的分析[J/OL]. 分子植物育种,2021:1-23[2021-09-01]. https://kns. cnki. net/kcms/detail/46.1068. s. 20210827.1325. 005. html. Li NY,Zhang HS,An HM,Lu M. Analysis of genetic diversity of Rosa roxburghii Tratt. based on ITS sequence[J/OL]. Molecular Plant Breeding,2021:1-23[2021-09-01]. https://kns.cnki.net/kcms/detail/46.1068.s.20210827.1325.005.html.
[24] 孟艺宏,徐刚标,卢孟柱,姜小龙,郭飞龙. 长柄双花木种群遗传结构及种群历史[J]. 林业科学,2020,56(7):55−62. Meng YH,Xu GB,Lu MZ,Jiang XL,Guo FL. Population genetic structure and demographic history of Disanthus cercidifolius var. longipes[J]. Scientia Silvae Sinicae,2020,56(7):55−62.
[25] 闫丽君. 基于SSR标记的伯乐树遗传多样性分析[D]. 长沙:中南林业科技大学,2015:19−28. [26] Li LX,Ou WL,Wang YC,Peng JY,Wang DW,Xu S. Comparison of genetic diversity between ancient and common populations of Docynia delavayi (Franch.) Schneid[J]. Gene,2022,829:146498.
[27] 吴敏,吴诗琪,潘凤,石甜,赵财. 中国野生半夏的遗传多样性和遗传结构研究[J/OL]. 广西植物,2023:1-13[2023-07-25]. https://kns. cnki. net/kcms2/detail/45.1134. q. 20230724.1642. 004. html. Wu M,Wu SQ,Pan F,Shi T,Zhao C. Genetic diversity and genetic structure of wild Pinellia ternate(Araceae) in China[J]. Guihaia,2023:1-13[2023-07-25]. https://kns.cnki.net/kcms2/detail/45.1134.q.20230724.1642.004.html.
[28] 张怀山. 基于SSR标记及ITS、cpDNA序列的刺梨遗传多样性分析[D]. 贵阳:贵州大学,2017:23−60. [29] 刘慎谔. 刘慎谔文集[M]. 北京:科学出版社,1985:23−100. [30] 吴诗琪,潘凤,赵财. 西南地区野生刺梨的遗传多样性和遗传结构研究[J]. 广西植物,2023,43(11):2065−2077. Wu SQ,Pan F,Zhao C. Genetic diversity and structure of wild Rosa roxburghii in Southwest China[J]. Guihaia,2023,43(11):2065−2077.
-
期刊类型引用(1)
1. 何拓,曾岩,殷亚方,张坤,袁良琛,董晖,周志华. 为野生植物保护和可持续贸易奠定科学基础——CITES植物委员会第27次会议评述. 生物多样性. 2024(09): 171-175 .  百度学术
百度学术
其他类型引用(0)



 下载:
下载: