Effects of salicylic acid on acteoside accumulation and gene expression in tuberous roots of Rehmannia glutinosa Libosch.
-
摘要:
为了分析叶片表面喷施水杨酸(SA)对毛蕊花糖苷含量的影响及其分子调控特征,以地黄(Rehmannia glutinosa Libosch.)栽培品种‘温85-5’为材料,用100 μmol/L的SA喷施生长180 d的植株,在处理0、1、3和6 h后收集叶片和块根,测定毛蕊花糖苷的含量,并对块根进行转录组测序分析。结果显示,SA处理后,地黄叶片中的毛蕊花糖苷提高了11.2% ~ 19.3%,块根中的毛蕊花糖苷含量提高了0.9 ~ 1.4倍。转录组分析结果表明,SA处理3 h后的差异表达基因最多,且下调表达的基因多于上调表达。苯乙醇苷生物合成通路在差异表达基因中得到显著富集,毛蕊花糖苷合成途径的催化酶基因如ALDH、UGT和PPO等在SA处理后的地黄块根中上调表达。此外,本文还鉴定出多个在SA处理后差异表达的AP2-EREBP、WRKY和MYB等转录因子基因。
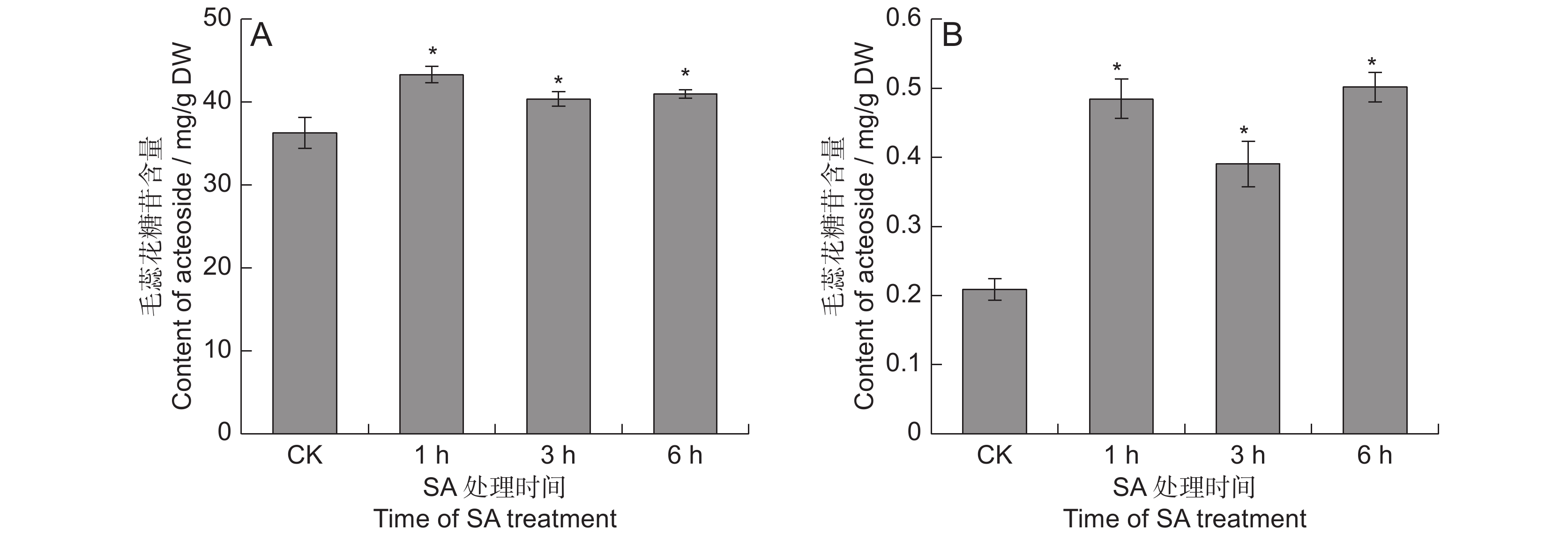
Abstract:The Rehmannia glutinosa ‘Wen 85-5’ cultivar was used to analyze the effects of spraying salicylic acid (SA) on the leaf surface on acteoside content and molecular regulation characteristics. The plants were grown for 180 days, then sprayed with SA (100 μmol/L). The leaves and tuberous roots of R. glutinosa were collected at 0, 1, 3, and 6 h after treatment to determine the content of acteoside. Transcriptome sequencing of the tuberous roots at different times after SA treatment was also performed. Results showed that, compared with the controls, acteoside content in the leaves and tuberous roots increased by 11.2% - 19.3%, and 0.9 - 1.4 times, respectively, after SA treatment. Transcriptome analysis showed that most differentially expressed genes (DEGs) were obtained 3 h after SA treatment, with more down-regulated genes than up-regulated genes. Most DEGs were significantly enriched in the phenylpropanoid biosynthesis pathway, while several catalytic enzyme genes of the acteoside synthesis pathway, such as ALDH, UGT, and PPO, were up-regulated in the tuberous roots. Many AP2-EREBP, WRKY, and MYB transcription factor genes were differentially expressed after SA treatment. This study provides theoretical support for the use of elicitors to treat R. glutinosa plants in the field to increase acteoside content.
-
Keywords:
- Rehmannia glutinosa /
- Salicylic acid /
- Foliar-spraying /
- Acteoside /
- Gene expression
-
生态位(Niche)是衡量物种所需环境资源的关键指标[1]。自Grinnell[2]在1917年定义生态位概念以来,其研究便成为生态学领域研究的热点。森林群落生态位研究中,生态位宽度、生态位重叠与生态位相似性作为3个关键的度量反映了群落内物种种间关系及与环境关系的动态变化[3]。生态位宽度作为衡量物种资源利用的重要指标,反映物种对环境资源的利用程度,也预示了其生态适应性与分布幅度[4];生态位重叠反映了物种在竞争或共享同级环境资源时的生态关系[5];生态位相似性则反映了不同物种在环境资源利用方面的相似性水平[6]。森林群落生态位研究中群落内部物种资源竞争及利用情况是研究的重要内容,其中在资源利用方面以随机生态位边界假说和生态位优先占领假说为主。随机生态位边界假说通过假定种的生态位多维体积的边界,各物种通过随机方式占据生态位的多维空间利用资源,根据物种在群落内相对重要性大小排布,依次形成一条平坦的曲线;生态位优先占领假说通过假定生态位多维空间的大小,主要由竞争能力较强的物种优先占领较大生态位,其余物种依次占据剩余生态位,各物种在群落内的相对重要性大小排布形成几何级数的陡直曲线[7]。探究群落生态位特征并对生态位空间理论的假说进行检验,对于深入理解自然群落内物种间的共存与竞争机制至关重要。
目前对于不同森林类型的群落生态位特征已开展大量的研究[5, 8-10],这些研究多基于优势种群生态位及各生态位指数间的相互关系,对理解森林优势种群物种维持机制有一定意义,但海拔梯度变化对生态位特征影响的研究鲜有报道[11-13]。鉴于物种生态位与环境间的复杂关系及区域间的特异性,针对不同地区开展环境梯度的变化对区域生态位特征的影响十分必要。
南岭地区作为连接南亚热带和中亚热带的生态过渡区域,保存了世界上同纬度地区最为完整的亚热带森林植被带谱系列[14],沿海拔梯度由低至高可见多种植被类型分布[15]。海拔作为一个综合性指标,综合了水分、温度、光照和土壤肥力等多种环境因素[16],对生物多样性及群落结构具有根本性影响。对此本研究提出两个科学问题:(1)各生态位特征指标间存在怎样的内在联系和相关性?(2)南岭中段森林优势种生态位垂直分布各符合何种生态位资源利用假说?通过对比分析不同海拔梯度下南岭中段地区森林优势树种各生态位指标,揭示优势树种组成及其生态位的变化规律,探讨海拔梯度对优势树种的影响机制,论证森林群落生态位空间理论,以期为南岭中段森林群落对于环境资源的响应研究以及南岭山地森林生态系统的可持续发展和植物物种多样性保护提供理论支持。
1. 研究区概况
本研究区域位于广东省南岭国家级自然保护区(24°37′~24°57′N,112°30′~113°04′E)。该区域属中亚热带湿润性季风气候,年平均温度17.7 ℃,多年平均降水量为1 705 mm[17]。区域的成土母岩以花岗岩、砂页岩和石灰岩为主[18]。随着海拔梯度变化,土壤类型依次呈现红壤、山地红黄壤、山地黄壤以及山地灌丛草甸土等[19]。森林植被资源丰富,主要植被类型为亚热带常绿阔叶林和针叶林[20]。随海拔高度递增,植被类型依次为沟谷常绿阔叶林(<800 m)、山地常绿阔叶林(800~<1 200 m)、针阔混交林(1 200~<1 500 m)、常绿阔叶矮林(≥1 500 m)[15]。
2. 研究方法
2.1 样地设置与调查方法
于2022年2-6月,在南岭中段海拔400~1 800 m,天然林保存完整的地段,按100 m海拔梯度,每个梯度3个重复,共设置植物样地45块。样地基本沿广东第一峰(海拔1 902 m)至南水水库(海拔212 m)一线设置。每个海拔梯度选择人为干扰较少且能代表该海拔段植被类型的天然林设置3个重复样地。样地采用长方形或正方形,最小边长>样地内平均树高的2.5倍。样地沿山体走向设置,不跨过道路河流,不跨不同林分,距离道路或林缘 >20 m,重复样地间距离 >30 m。样地采用PVC管和尼龙绳分割成10 m×10 m的小样方以方便调查,PVC管放样时采用RTK确定位置,放样误差控制在 <0.1 m。根据南岭地区沿海拔梯度植被类型的不同将15个梯度等级归类为4个森林植被类型(表1),分别为沟谷常绿阔叶林(300~700 m)、山地常绿阔叶林(800~1 100 m)、针阔混交林(1 200~1 500 m)和常绿阔叶矮林(1 600~1 800 m)。在所选样地范围内,对所有胸径在1 cm及以上的木本植物进行精确的地理位置标记,并鉴定其物种名称。调查其胸径、树高、冠幅等关键生长参数,记录该样地的海拔、经纬度以及坡位坡向等环境特征。
表 1 样地基本信息Table 1. Basic information of sample plots样地编号
Sampling site面积
Area / m2海拔
Altitude / m坡度
Slope / °坡向
Aspe林分起源
Stand origin350-1 1 200 342 10 东 次生林 350-2 1 200 344 10 东 次生林 350-3 1 200 337 6 东 次生林 500-1 1 200 494 28 西南 次生林 500-2 1 200 526 35 南 次生林 500-3 1 200 530 30 西南 次生林 600-1 1 200 619 22 南 次生林 600-2 1 200 604 28 南 次生林 600-3 1 200 605 30 东南 次生林 700-1 1 200 703 18 西北 次生林 700-2 1 200 708 22 东南 次生林 700-3 1 200 712 20 东南 次生林 800-1 1 200 810 20 西南 次生林 800-2 1 200 792 18 东南 次生林 800-3 1 200 785 16 东南 次生林 900-1 1 200 900 15 南 次生林 900-2 1 200 904 12 南 次生林 900-3 1 200 885 15 南 次生林 1000-1 1 200 1 026 13 东南 次生林 1000-2 1 200 1 013 18 东南 次生林 1000-3 1 200 1 002 20 东南 次生林 1100-1 1 200 1 112 18 西 次生林 1100-2 1 200 1 109 20 西 次生林 1100-3 1 200 1 103 20 西 次生林 1200-1 1 200 1 210 26 西南 次生林 1200-2 1 200 1 200 22 西南 次生林 1200-3 1 200 1 202 22 西南 次生林 1300-1 1 200 1 350 25 东南 次生林 1300-2 1 200 1 297 23 东 次生林 1300-3 1 200 1 345 30 东南 次生林 1400-1 1 200 1 462 35 东南 原生林 1400-2 1 200 1 432 30 东南 原生林 1400-3 1 200 1 395 26 东北 原生林 1500-1 900 1 558 5 西南 原生林 1500-2 900 1 526 32 西南 原生林 1500-3 900 1 482 35 西南 原生林 1600-1 1 200 1 622 20 西南 原生林 1600-2 1 200 1 628 22 西南 原生林 1600-3 1 200 1 648 30 西南 原生林 1700-1 400 1 700 6 西南 原生林 1700-2 400 1 705 6 西南 原生林 1700-3 400 1 710 6 西南 原生林 1800-1 400 1 778 30 东 原生林 1800-2 400 1 789 28 东 原生林 1800-3 400 1 792 25 南 原生林 2.2 数据处理与统计分析
2.2.1 物种重要值及变异系数
重要值计算[21]:
IV = (RA + RF + RD)/3×100% 式中,IV为物种重要值,RA、RF和RD分别为相对多度、相对频度和相对显著度。
物种重要值变异系数计算[12]:
CV=SD/M 式中,CV为物种重要值变异系数,SD为各物种重要值的标准差,M为各物种重要值的平均值。
2.2.2 生态位宽度
生态位宽度分别采用Levins生态位宽度(BL)[22]和Shannon-Wiener 生态位宽度(BSW)[23]来计算:
BL=1/r∑j=1P2ij Bsw=−r∑j=1Pij⋅lnPij 式中,Pij=nij/Nij,Nij为物种i在所有资源位重要值总和,nij为物种i在资源位j的重要值,r为样方数。
2.2.3 生态位重叠指数
生态位重叠采用Pianka[24]生态位重叠度公式来计算:
Oih=r∑j=1PijPhj/√r∑j=1P2ijr∑j=1P2hj 式中,Oih为物种i和物种h的生态位重叠度指数。
2.2.4 生态位相似指数
生态位相似指数采用Cih计算公式计算[25]:
Cih=1−12r∑j=1|Pij−Phj| 式中,Cih为物种i和物种h的生态位相似性指数。
2.2.5 数据处理
分别采用Excel 2016和R4.1.0中的spaa包进行计算物种重要值和生态位宽度、生态位重叠指数、生态位相似指数。同时使用R软件中的vegan包进行非度量多维尺度分析(NMDS)和典范对应分析(CCA)排序。
3. 结果与分析
3.1 不同海拔梯度优势树种重要值及其变异性
根据各群落类型特征,15个海拔梯度群落可划分为4个植被类型:即沟谷常绿阔叶林、山地常绿阔叶林、针阔混交林和常绿阔叶矮林。45块植物样地共记录乔木树种263种,根据物种重要值排序选取前15的树种作为该区域的优势树种进行生态位特征分析。从表2中可以看出,所有样地内各树种重要值由大到小排序依次为:甜槠(Castanopsis eyrei (Champ.) Tutch)、青冈(Cyclobalanopsis glauca (Thunb.) Oerst)、木荷(Schima superba Gardn. et Champ.)、小红栲(Castanopsis carlesii (Hemsl.) Hay)、木莲(Manglietia fordiana Oliv. in Hook.)、杉木(Cunninghamia lanceolata (Lamb.) Hook.)、拟赤杨(Alniphyllum fortunei (Hemsl.) Makino)、华南五针松(Pinus kwangtungensis Chun ex Tsiang)、华润楠(Machilus chinensis (Champ. ex Benth.) Hemsl. )、杨桐(Adinandra millettii Hook. et Arn.)、五列木 (Pentaphylax euryoides Gardn. et Champ.)、罗浮柿(Diospyros morrisiana Hance)、红楠(Machilus thunbergii Sieb. et Zucc.)、假地枫皮(Illicium jiadifengpi B. N. Chang)、尖叶柃(Eurya acuminatissima Merr. et Chun)。其中沟谷常绿阔叶林物种重要值较大的树种有:甜槠(10.8%)、青冈(8.3%)和木荷(4.8%);山地常绿阔叶林物种重要值较大的树种为:小红栲(10.1%)、甜槠(9.0%)和木荷(8.8%);针阔混交林物种重要值较大的树种为:华南五针松(10.5%)、木荷(6.5%)和青冈(5.9%);常绿阔叶矮林物种重要值较大的树种为:假地枫皮(13.6%)、青冈(13.3%)和木莲(5.9%)。物种重要值变异系数较大的树种分别为假地枫皮(2.775)、华南五针松(2.700)和杉木(1.625),以上几种树种多集中分布同一植被类型内;物种变异系数较小的树种分别为杨桐(0.793)、甜槠(0.813)和华润楠(0.840),以上树种在各海拔梯度内重要值差异不大且每个海拔梯度内均有一定分布。在所有样地中重要值最高的3个树种甜槠、青冈和木荷其变异系数值均较低,表明甜槠、青冈和木荷在南岭地区各海拔梯度分布范围广泛且在其所属森林群落中具有高优势度,是南岭地区较有代表性的树种。通过NMDS分析对不同海拔梯度优势种差异性进行分析(图1),得出优劣用胁强系数(Stress)为0.1822,排序分析结果合理。同时其P值为0.001,可知不同海拔梯度内优势树种组成差异显著。
表 2 不同海拔优势树种重要值及其变异系数Table 2. Importance values and coefficients of variation of dominant tree species at different altitudes代码
No.重要值Importance value / % 变异系数
Variance coefficient沟谷常绿阔叶林
Valley evergreen
broad-leaved forest山地常绿阔叶林
Montane broad-leaved evergreen forest针阔混交林
Coniferous and
broad-leaved mixed forest常绿阔叶矮林
Evergreen broad-leaved
dwarf forest所有样地
All plotsS1 10.8 9.0 5.3 5.1 9.3 0.813 S2 8.3 2.1 5.9 13.3 8.0 0.971 S3 4.8 8.8 6.5 3.3 7.9 0.845 S4 4.8 10.1 0.1 − 4.9 1.531 S5 1.6 5.6 2.4 5.9 4.8 1.125 S6 4.0 7.9 1.1 − 4.3 1.625 S7 3.5 5.1 2.1 0.1 4.1 0.951 S8 − − 10.5 − 4.0 2.700 S9 3.2 3.4 2.9 0.4 3.8 0.840 S10 2.3 3.4 2.7 0.5 3.6 0.793 S11 0.9 1.8 5.6 0.2 3.3 1.365 S12 3.7 3.8 0.3 − 3.1 1.157 S13 3.2 2.7 2.2 0.2 3.1 1.381 S14 0.1 0.1 0.9 13.6 3.0 2.775 S15 1.5 2.4 2.7 0.3 2.8 1.040 注:S1,甜槠;S2,青冈;S3,木荷;S4,小红栲;S5,木莲;S6,杉木;S7,拟赤杨;S8,华南五针松;S9,华润楠;S10,杨桐;S11,五列木;S12,罗浮柿;S13,红楠;S14,假地枫皮;S15,尖叶柃。下同。 Notes: S1, Castanopsis eyrei (Champ.) Tutch; S2, Cyclobalanopsis glauca (Thunb.) Oerst; S3, Schima superba Gardn. et Champ.; S4, Castanopsis carlesii (Hemsl.) Hay; S5, Manglietia fordiana Oliv. in Hook.; S6, Cunninghamia lanceolata (Lamb.) Hook.; S7, Alniphyllum fortunei (Hemsl.) Makino; S8, Pinus kwangtungensis Chun ex Tsiang; S9, Machilus chinensis (Champ. ex Benth.) Hemsl.; S10, Adinandra millettii Hook. et Arn.; S11, Pentaphylax euryoides Gardn. et Champ.; S12, Diospyros morrisiana Hance; S13, Machilus thunbergii Sieb. et Zucc.; S14, Illicium jiadifengpi B. N. Chang; S15, Eurya acuminatissima Merr. et Chun. Same below. 3.2 不同海拔梯度优势树种生态位宽度
由表3可知,随着海拔梯度的变化优势树种的生态位宽度表现出差异性。在所有样地中,Levins生态位宽度计算排序前5的树种分别为杨桐(27.856)、甜槠(27.338)、华润楠(26.627)、木荷(26.502)和拟赤杨(23.877);Shannon-Wiener生态位宽度计算排序前5的树种为杨桐(3.419)、木荷(3.412)、青冈(3.412)、甜槠(3.403)和华润楠(3.384),两种生态位宽度计算方法所得生态位宽度较高的物种高度一致,揭示了这些树种在不同海拔梯度上普遍分布,并且对环境变化具有较强适应性。Levins和Shannon-Wiener生态位宽度值排序最后的物种均为杉木、华南五针松和假地枫皮,其数值分别为12.565和2.662 ;5.535和1.794;5.276和2.054,说明这3个树种对该区的环境资源能力相对较弱,数量较少或分布相对集中,导致其生态位宽度也较小。沟谷常绿阔叶林区域生态位宽度较大的树种分别为罗浮柿、甜槠和拟赤杨;山地常绿阔叶林区域生态位宽度较大的树种分别为杨桐、拟赤杨、华润楠和青冈;针阔混交林区域生态位宽度较大的树种分别为杨桐、甜槠、华润楠和青冈;常绿阔叶林区域生态位宽度较大的树种分别为青冈、木莲和假地枫皮。说明不同海拔梯度植被优势种的生态位宽度存在差异,尤其是沟谷常绿阔叶林和常绿阔叶矮林之间差异更为明显。
表 3 不同海拔梯度优势树种生态位宽度Table 3. Niche breadth of dominant species at different altitudinal gradients代码
No.沟谷常绿阔叶林
Valley evergreen broad-leaved forest山地常绿阔叶林
Montane broad-leaved evergreen forest针阔混交林
Coniferous and broad-leaved mixed forest常绿阔叶矮林
Evergreen broad-leaved dwarf forest所有样地
All plotsBL Bsw BL Bsw BL Bsw BL Bsw BL Bsw S1 7.951 2.193 8.034 2.191 8.149 2.182 2.886 1.079 27.338 3.403 S2 6.866 2.136 9.043 2.296 7.965 2.215 4.593 1.753 23.416 3.412 S3 7.631 2.140 8.576 2.303 7.431 2.157 2.906 1.083 26.502 3.412 S4 7.252 2.137 7.279 2.107 1.972 0.686 − − 13.666 2.805 S5 5.429 1.852 6.047 1.990 6.035 1.990 4.577 1.730 20.112 3.239 S6 2.662 1.080 7.720 2.143 2.311 1.151 − − 12.565 2.662 S7 7.647 2.244 9.485 2.361 5.757 1.922 1.996 0.692 23.877 3.327 S8 − − − − 5.535 1.794 − − 5.535 1.794 S9 7.580 2.177 9.238 2.260 7.996 2.193 2.882 1.077 26.627 3.384 S10 6.399 1.974 10.351 2.395 9.216 2.302 2.885 1.078 27.856 3.419 S11 5.583 1.851 7.511 2.166 7.386 2.184 1.000 0.000 15.942 3.060 S12 9.852 2.357 8.455 2.234 1.609 0.695 − − 19.483 3.070 S13 5.316 1.795 6.756 2.117 3.357 1.382 1.000 0.000 15.712 2.943 S14 2.651 1.037 2.000 0.693 2.528 1.346 3.662 1.498 5.276 2.054 S15 5.310 1.856 8.499 2.266 7.059 2.047 2.709 1.040 21.880 3.236 通过对研究区域45个样地进行典范对应分析(CCA)得到生态位宽度与各环境因子二维排序图(图2)。3个环境因子海拔、坡度、坡向对优势种生态位宽度解释量为10.79%,第1排序轴与第2排序轴占排序轴特征值的10.11%,同时,显著性检验结果表明海拔是最优预测因子,进一步说明了海拔是影响研究区优势种生态位宽度的重要环境因子之一。
3.3 不同海拔梯度优势树种生态位重叠
从图3可以看出,不同海拔优势种生态位重叠明显的物种对存在一定差异,沟谷常绿阔叶林(图3:A)中生态位重叠度较大的树种对为拟赤杨-尖叶柃(0.93)、小红栲-红楠(0.90)和甜槠-罗浮柿(0.88);山地常绿阔叶林(图3:B)海拔梯度生态位重叠度较大的树种对为甜槠-青冈(0.91)、杨桐-杉木(0.90)和拟赤杨-罗浮柿(0.89);针阔混交林(图3:C)海拔梯度生态位重叠度较大的树种对为尖叶柃-华润楠(0.93)、杨桐-华润楠(0.91)和杨桐-尖叶柃(0.87);常绿阔叶矮林(图3:D)海拔梯度为五列木-红楠(0.97)、木荷-假地枫皮(0.96)和木莲-假地枫皮(0.96);若将区域扩大为所有海拔(图3:E),生态位重叠度最大的树种对是杨桐-尖叶柃(0.81)、拟赤杨-罗浮柿(0.80)和杨桐-华润楠(0.80)。以上树种对在不同海拔生境需求具有相似性,种间竞争激烈。
![]() 图 3 不同海拔梯度优势种生态位重叠指数A:沟谷常绿阔叶林;B:山地常绿阔叶林;C:针阔混交林;D:常绿阔叶矮林:E:所有海拔梯度。图中指数数值越大,圆圈越大,颜色向深红色渐变;反之,颜色向深蓝色渐变。下同。Figure 3. Ecological niche overlap index of dominant species at different altitudinal gradientsA: Valley evergreen broad-leaved forest; B: Montane broad-leaved evergreen forest; C: Coniferous and broad-leaved mixed forest; D: Evergreen broad-leaved dwarf forest; E: All elevation gradients. Larger index value indicates larger circle, and color gradually changes to dark red; in contrast, color gradually changes to dark blue. Same below.
图 3 不同海拔梯度优势种生态位重叠指数A:沟谷常绿阔叶林;B:山地常绿阔叶林;C:针阔混交林;D:常绿阔叶矮林:E:所有海拔梯度。图中指数数值越大,圆圈越大,颜色向深红色渐变;反之,颜色向深蓝色渐变。下同。Figure 3. Ecological niche overlap index of dominant species at different altitudinal gradientsA: Valley evergreen broad-leaved forest; B: Montane broad-leaved evergreen forest; C: Coniferous and broad-leaved mixed forest; D: Evergreen broad-leaved dwarf forest; E: All elevation gradients. Larger index value indicates larger circle, and color gradually changes to dark red; in contrast, color gradually changes to dark blue. Same below.各个海拔梯度等级生态位重叠指数平均值的排序依次为:常绿阔叶矮林(0.72)>山地常绿阔叶林(0.62)>沟谷常绿阔叶林(0.53)>针阔混交林(0.44),常绿阔叶矮林区域优势树种对资源环境的需求具有较高的相似性,从而导致了更为激烈的种间竞争;相比之下,针阔混交林区域优势树种对资源环境需求的相似程度较低。在所有海拔梯度区域内,乔木优势种共组成了105组种对,生态位重叠指数平均值为0.45,其中生态位重叠小于0.3的种对有23对(21.90%),在0.3~0.6的种对有58对(55.24%),大于0.6的种对有24对(22.86%),不发生重叠的种对有2对(1.9%),分别为小红栲-华南五针松和杉木-假地枫皮。所有样地内各优势种树种对之间普遍存在生态位重叠,结果揭示了该区域的树种在利用环境资源方面表现出较高的适应性,种间关系协调。
3.4 不同海拔梯度优势树种生态位相似性
从图4可以看出,不同海拔梯度优势树种生态位相似度较大的物种对存在一定的差异,沟谷常绿阔叶林(图4:A)生态位相似性较高的树种对为木荷-甜槠(0.77)、尖叶柃-拟赤杨(0.76)和华润楠-罗浮柿(0.75);山地常绿阔叶林(图4:B)生态位相似性较高的树种对为青冈-甜槠(0.83)、杉木-杨桐(0.78)和罗浮柿-拟赤杨(0.76);针阔混交林(图4:C)生态位相似性较高的树种对为华润楠-尖叶柃(0.84)、华润楠-杨桐(0.78)和尖叶柃-杨桐(0.75);常绿阔叶矮林(图4:D)生态位相似性较高的树种对为红楠-五列木(0.97)、木荷-甜槠(0.97)和假地枫皮-木荷(0.88);若将区域扩大为所有海拔(图4:E),生态位相似性最高的树种对是华润楠-杨桐(0.73)、木荷-甜槠(0.70)、华润楠-拟赤杨(0.69)和尖叶柃-杨桐(0.69)。说明以上物种对在各海拔梯度内,对资源的利用程度和竞争能力相较其他物种对更为接近。
在不同的海拔梯度内生态位相似性均值大小排序为:常绿阔叶矮林(0.54)>山地常绿阔叶林(0.53)>沟谷常绿阔叶林(0.46)>针阔混交林(0.38),这表明常绿阔叶矮林海拔梯度优势树种对生态需求更为接近,在所有海拔梯度区域内,乔木优势种共组成了105组种对,生态位相似性均值为0.39,其中生态位重叠小于0.3的种对有26对(24.76%),在0.3~0.6的种对有67对(63.81%),大于0.6的种对有12对(11.43%),多数树种对生态位相似性指数围绕所有种对的均值波动,说明该区各树种对区域内资源利用较为充分。
3.5 重要值及其变异系数与生态位宽度的关系
由图5可知所有海拔梯度优势树种Levins和Shannon-Wiener生态位宽度值与重要值之间呈微弱正相关,其Pearson相关系数分别为0.445和0.384。由图6可知所有海拔梯度优势树种生态位宽度值与重要值变异系数之间呈显著负相关,其Pearson相关系数分别为−0.953和−0.986,说明在本研究中优势种的重要值与生态位宽度关系不密切,而重要值变异系数与其生态位宽度成反比,即变异系数增大时生态位宽度减小,反之则增大。
4. 讨论
4.1 重要值和生态位宽度
物种重要值能反映物种在群落中的优势度;物种的生态位宽度则反映物种在环境中的适应力与种间竞争力[26]。在以往的研究中,物种重要值和生态位宽度存在一定的关联性且多表现为正相关[8, 27],本研究中,优势树种重要值与生态位宽度之间呈微弱正相关,且多种树种的重要值与生态位宽度值排列位置并不相称,如重要值排位第1和2位的甜槠和青冈,其生态位宽度排位在第2和6位,这与李一凡等[5]研究结果一致。这可能是因为影响物种生态位宽度的因素较为复杂,且本研究通过对南岭中段森林优势树种生态位宽度进行典范对应分析(CCA)发现其对海拔、坡向和坡位3个环境因子分析所得解释量不高,证明还有许多本研究未调查的环境因子对该地区优势种生态位宽度有影响,结合以往的研究[11, 28],发现生态位宽度与土壤pH值、土壤全磷、全钾、枯枝落叶层、腐殖质层厚度和林冠郁闭度等均有关,在后续的研究中可以增加更多维的环境因子加以分析。此外,该区群落多为天然次生林,未达到演替顶级阶段,生态位宽度也没有到最大。重要值变异系数作为重要值在不同样地间离散程度的量度,与生态位宽度之间呈现显著的负相关,这与前人的研究结果一致[25, 29],这可能与重要值离散程度某种程度上反映物种适应环境的能力有关。因此,重要值和生态位宽度虽然作为物种生态位特征的重要评价指标,但不能单纯以某种指标作为判断物种生态位综合特征的依据[30]。
4.2 生态位重叠与相似性
生态位的重叠通常是物种间竞争和相互作用的指标[31]。一般认为,每个种对生态位重叠越大,两个物种之间竞争越强烈[32],但也有研究表明,仅在资源匮乏的地区才会引起竞争[33]。在本研究中,随着海拔梯度变化,不同优势种间的生态位重叠度存在明显差异,在最低和最高海拔梯度生态位重叠度相较于中间海拔梯度整体较高,这可能是由于最低海拔虽然水热条件好,但物种数量也多,对资源的需求量大,而高海拔自然条件恶劣[34],资源的限制导致竞争激烈;中间海拔生态位重叠低符合生物多样性“中间膨胀”理论[35],温度和能量随海拔的升高而降低,水分随海拔的升高而升高,因此,在三者共同作用下中海拔段形成最适物种分布区[36]。生态位相似性指标揭示了物种在对共有资源的需求和利用上的相似性[37]。物种间相似性的变化趋势与生态位重叠的变化趋近,4个群落类型中处于中高海拔的针阔混交林值最低,群落稳定性最高[12]。
根据以往的研究成果,生态位宽度与生态位重叠及相似性之间多为正相关[38-40],少数为无明显关联[41],甚至生态位宽度大的种对间也可能具有较小的生态位相似性[42],从本研究结果来看,生态位宽度值较大树种之间可能产生较高的生态位相似性和生态位重叠值,如甜槠和青冈,但较小生态位宽度值的树种与较大生态位宽度值的树种往往只有小的生态位重叠值,如小红栲与华南五针松,而较小生态位宽度值的树种之间则可有较高的生态位重叠值,也可有较大的生态位相似性比例,如尖叶柃与假地皮枫,这与闫东锋等[12]对太行山南麓不同海拔梯度天然林优势树种生态位的研究结果一致。总的来说,生态位宽度与生态位重叠指数及生态位相似性指数之间的相互作用是复杂的,并且与物种对的生态行为、生境因子和采样尺度紧密相关[6]。
4.3 生态位资源利用假说验证
生态位资源利用假说是一种关于植物在群落中利用空间资源方式的理论假设。本研究中,处于中低海拔区域内的各优势树种重要值分布比较均匀,同时研究结果中生态位相似指数也反映了其相较于高海拔的常绿阔叶矮林海拔区域更具有群落稳定性[6, 12, 41];徐卫等[43]在南岭地区开展沿海拔梯度植物群落物种多样性的研究也发现南岭地区乔木层α多样性随海拔升高呈现“单峰曲线”的变化趋势,中低海拔相较高海拔地区树种组成更丰富,各树种的生态位多维体积边界位于多维空间的随机位置,形成了多样化的树种组成方式,故中低海拔区域的林分对资源利用方式的划分较符合随机生态位边界假说;高海拔的常绿阔叶矮林优势树种青冈和假地枫皮重要值明显高出其余优势树种,由于高海拔区域生境条件更为严酷,青冈和假地枫皮长期适应形成的生存方式和竞争能力占据了较大的生态位空间,进而挤占了其他物种的环境资源,故高海拔区域的林分对资源利用方式符合生态位优先占领假说。
-
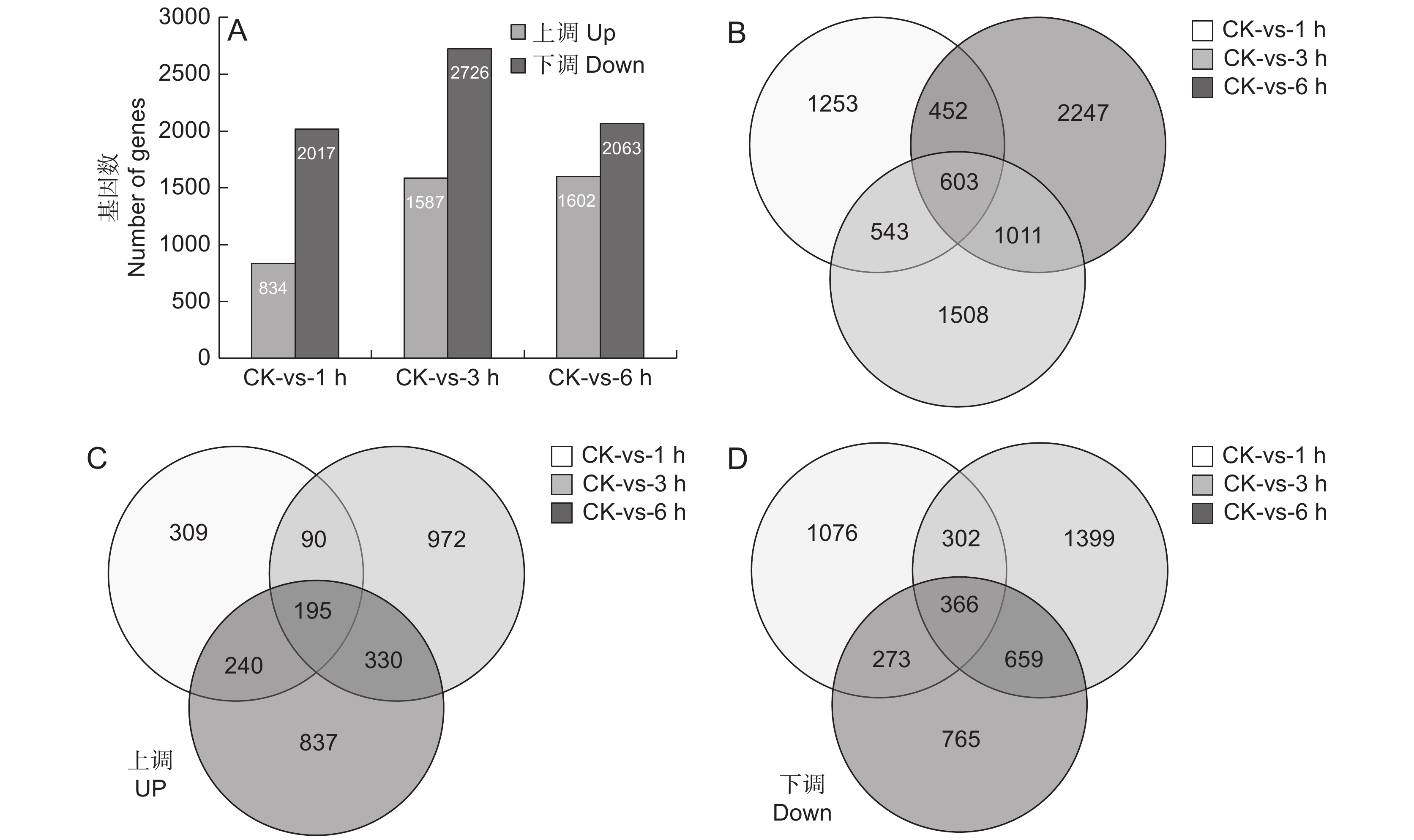
图 2 SA处理后基因显著差异表达
A:SA处理过程中上调和下调的基因数目; B ~ D:SA处理后不同时间点鉴别出的总DGEs(B)、上调DGEs(C)和下调DGEs的维恩图。
Figure 2. Significant DEGs in response to SA treatment
A: Up-regulated and down-regulated gene numbers during SA treatment; B − D: Venn diagram of total DEGs (B), up-regulated DEGs (C), and down-regulated DEGs (D) identified at different time points after SA treatment.
图 3 SA处理后3种比对均差异表达基因的GO分类及KEGG通路富集
A:603个基因的GO分类;B:KEGG富集的前20个代谢通路。圆点大小和颜色分别表示通路中DEGs的数量和Q值范围。1:分子转导活性;2:分子载体活性;3:信号转导活性;4:结构分子活性;5:转运活性;6:转录调节活性;7:抗氧化活性;8:结合;9:分子功能调节;10:催化活性;11:膜封闭腔;12:超分子复合物;13:共质体;14:细胞连接;15:细胞组分;16:细胞外区域;17:膜组分;18:膜;19:细胞器组分;20:细胞器;21:大分子复合物;22:细胞;23:生殖过程;24:繁殖;25:解毒作用;26:多细胞生物过程;27:发育过程;28:多生物体过程;29:信号;30:定域化;31:生物调节;32:对刺激的反应;33:代谢过程;34:细胞过程;35:细胞成分组织或生物合成。
Figure 3. GO classification and KEGG pathway enrichment of co-DEGs in three comparisons
A: GO classification of 603 genes; B: Top 20 enriched KEGG pathways among 603 genes. Size and color of dot represent number and scope of DEGs in pathway, respectively. 1: Molecular transducer activity; 2: Molecular carrier activity; 3: Signal transducer activity; 4: Structural molecule activity; 5: Transporter activity; 6: Transcription regulator activity; 7: Antioxidant activity; 8: Binding; 9: Molecular function regulator; 10: Catalytic activity; 11: Membrane-enclosed lumen; 12: Supramolecular complex; 13: Symplast; 14: Cell junction; 15: Cell part; 16: Extracellular region; 17: Membrane part; 18: Membrane; 19: Organelle part; 20: Organelle; 21: Macromolecular complex; 22: Cell; 23: Reproductive process; 24: Reproduction; 25: Detoxification; 26: Multicellular organismal process; 27: Developmental process; 28: Multi-organism process; 29: Signaling; 30: Localization; 31: Biological regulation; 32: Response to stimulus; 33: Metabolic process; 34: Cellular process; 35: Cellular component organization or biogenesis.
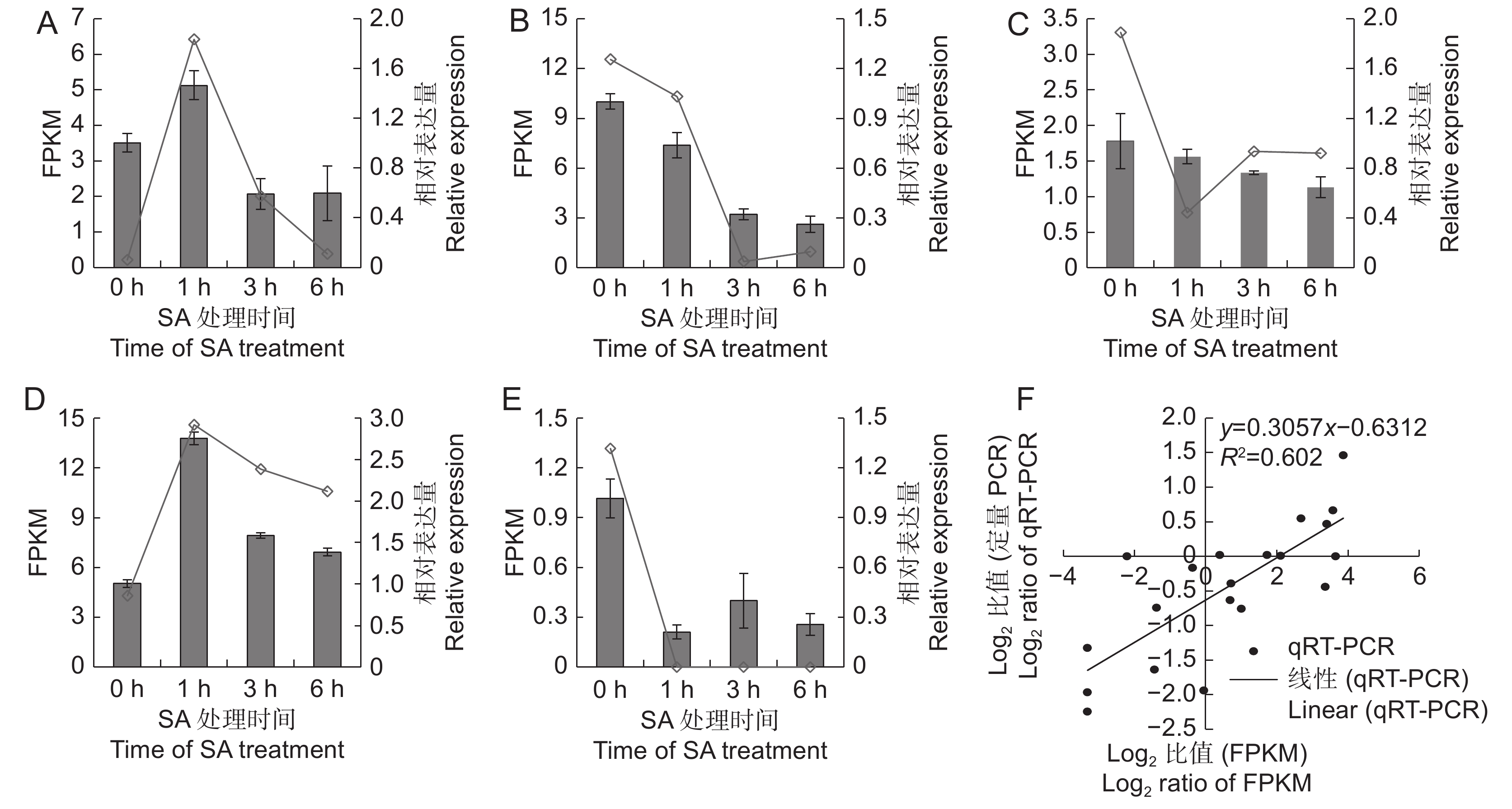
图 5 差异表达基因的qRT-PCR验证
A ~ E分别为CL7331.Contig2、Unigene1886、CL985.Contig1、CL5931.Contig2和CL379.Contig3的FPKM值与相对表达量;F:FPKM值与qRT-PCR的相关性分析。
Figure 5. Validation of DEGs by qRT-PCR
A–E: Represent expression and FPKM values of CL7331.Contig2, Unigene1886, CL985.Contig1, CL5931.Contig2, and CL379.Contig3; F: Correlation analysis between FPKM and qRT-PCR data.
表 1 测序数据统计结果
Table 1 Statistics of sequenced data
样本
Sample总原始序列
Total raw reads / M总测序序列
Total clean reads / M总测序碱基数
Total clean bases / Gb测序序列比率
Clean read ratio / %总匹配率
Total mapped / %特异匹配率
Uniquely mapped / %Control_1 21.94 21.13 1.06 96.3 87.70 51.31 Control_2 21.94 21.07 1.05 96.04 86.92 51.18 Control_3 21.94 21.08 1.05 96.08 85.72 51.09 SA 1h_1 21.94 21.27 1.06 96.93 85.44 47.35 SA 1h_2 21.94 21.08 1.05 96.05 87.07 48.03 SA 1h_3 21.94 21.15 1.06 96.37 87.28 49.26 SA 3h_1 21.94 20.98 1.05 95.59 86.85 50.36 SA 3h_2 21.94 21.05 1.05 95.93 85.74 49.92 SA 3h_3 21.94 21.05 1.05 95.95 86.81 50.47 SA 6h_1 21.94 21.09 1.05 96.11 86.50 50.25 SA 6h_2 21.94 21.06 1.05 96.00 85.04 49.42 SA6h_3 21.94 21.03 1.05 95.86 86.68 49.97 表 2 SA处理后差异表达的转录因子数
Table 2 Number of differentially expressed transcription factors (TFs) after SA treatment
转录因子
Transcription factorCK-vs-1 h CK-vs-3 h CK-vs-6 h 共同差异表达的基因数
Number of common DEGs下调 Down 上调 Up 下调 Down 上调 Up 下调 Down 上调 Up zf-HD 2 1 1 1 0 0 0 WRKY 11 5 8 16 5 15 5 TUB 1 0 1 1 0 0 0 Trihelix 1 0 1 0 0 0 0 Tify 1 0 3 0 3 0 1 PLATZ 0 4 0 4 1 3 2 SRS 0 0 2 0 2 0 0 OFP 2 0 3 0 1 0 0 NAC 2 8 2 6 0 8 4 MYB 15 10 5 7 6 12 3 mTERF 1 0 2 0 1 1 0 MADS 2 2 3 4 2 2 1 LOB 1 0 4 2 2 0 0 HSF 5 2 4 6 3 6 2 GRAS 1 8 0 4 0 7 2 G2-like 2 0 2 1 0 3 0 CPP 2 0 2 0 4 0 2 C2H2 3 1 8 1 3 1 2 C2C2-GATA 2 0 1 0 5 0 0 C2C2-Dof 1 1 3 0 2 1 0 C2C2-CO-like 0 1 0 2 4 3 1 bZIP 1 2 0 0 1 0 0 bHLH 12 3 15 4 5 4 2 AP2-EREBP 15 12 12 18 9 21 6 ABI3VP1 5 2 7 1 1 1 0 总数 88 62 89 78 60 88 33 表 3 SA处理后上调表达的转录因子基因
Table 3 Up-regulated transcription factor genes after SA treatment
转录因子
Transcription factor基因
GeneLog2(SA/CK) 功能
Function1 h 3 h 6 h AP2-EREBP CL1637.Contig3 2.82 1.29 2.42 Ethylene-responsive transcription factor ERF071 AP2-EREBP CL4501.Contig2 1.66 1.75 2.16 Pathogenesis-related genes transcriptional activator PTI6 AP2-EREBP CL7827.Contig1 1.77 3.51 4.24 Ethylene-responsive transcription factor ERF106-like AP2-EREBP Unigene2558 1.62 1.06 1.74 Ethylene-responsive transcription factor 2-like MYB CL1983.Contig1 4.55 4.19 3.12 Transcription factor TFIIIB component B''-like MYB CL4303.Contig1 3.76 2.61 3.47 Single MYB histone protein NAC CL2945.Contig2 6.82 7.15 7.51 NAC domain-containing protein 82-like isoform X1 NAC CL4851.Contig1 3.15 1.99 2.44 NAC transcription factor 29 NAC CL4851.Contig2 3.15 2.27 2.82 NAC transcription factor 29 NAC Unigene5193 2.09 1.38 2.37 NAC domain-containing protein 72 C2C2-CO-like CL5505.Contig3 2.94 2.66 4.31 Zinc finger protein CONSTANS-LIKE 4-like PLATZ CL5569.Contig1 2.31 1.49 2.53 Interleukin-1 receptor-associated kinase 4 PLATZ CL5569.Contig3 2.47 1.71 2.38 Interleukin-1 receptor-associated kinase 4 GRAS CL645.Contig2 1.93 1.65 2.91 Scarecrow-like protein 14 GRAS Unigene10453 1.03 1.03 1.08 Scarecrow-like protein 15 WRKY CL6521.Contig1 2.84 1.66 2.35 Probable WRKY transcription factor 25 WRKY CL7324.Contig3 1.13 1.59 1.21 Probable WRKY transcription factor 35 WRKY CL791.Contig6 2.59 2.28 2.29 Probable WRKY transcription factor 40 bHLH Unigene12420 2.20 2.20 2.29 Phytochrome-interacting factor 3 MADS Unigene23315 2.17 3.01 2.27 MADS-box transcription factor -
[1] 解杨,钟凌云,王卓,宋金菊,李家晴,等. 地黄炮制历史沿革及现代研究进展[J]. 中国实验方剂学杂志,2022,28(2):273−282. Xie Y,Zhong LY,Wang Z,Song JJ,Li JQ,et al. Historical evolution and modern research progress of Rehmanniae radix[J]. Chinese Journal of Experimental Traditional Medical Formulae,2022,28 (2):273−282.
[2] 国家药典委员会. 中华人民共和国药典: 一部[M]. 北京: 中国医药科技出版社, 2020: 129−130. [3] 陈金鹏,张克霞,刘毅,盖晓红,任涛,等. 地黄化学成分和药理作用的研究进展[J]. 中草药,2021,52(6):1772−1784. doi: 10.7501/j.issn.0253-2670.2021.06.028 Chen JP,Zhang KX,Liu Y,Gai XH,Ren T,et al. Research progress on chemical constituents and pharmacological actions of Rehmannia glutinosa[J]. Chinese Traditional and Herbal Drugs,2021,52 (6):1772−1784. doi: 10.7501/j.issn.0253-2670.2021.06.028
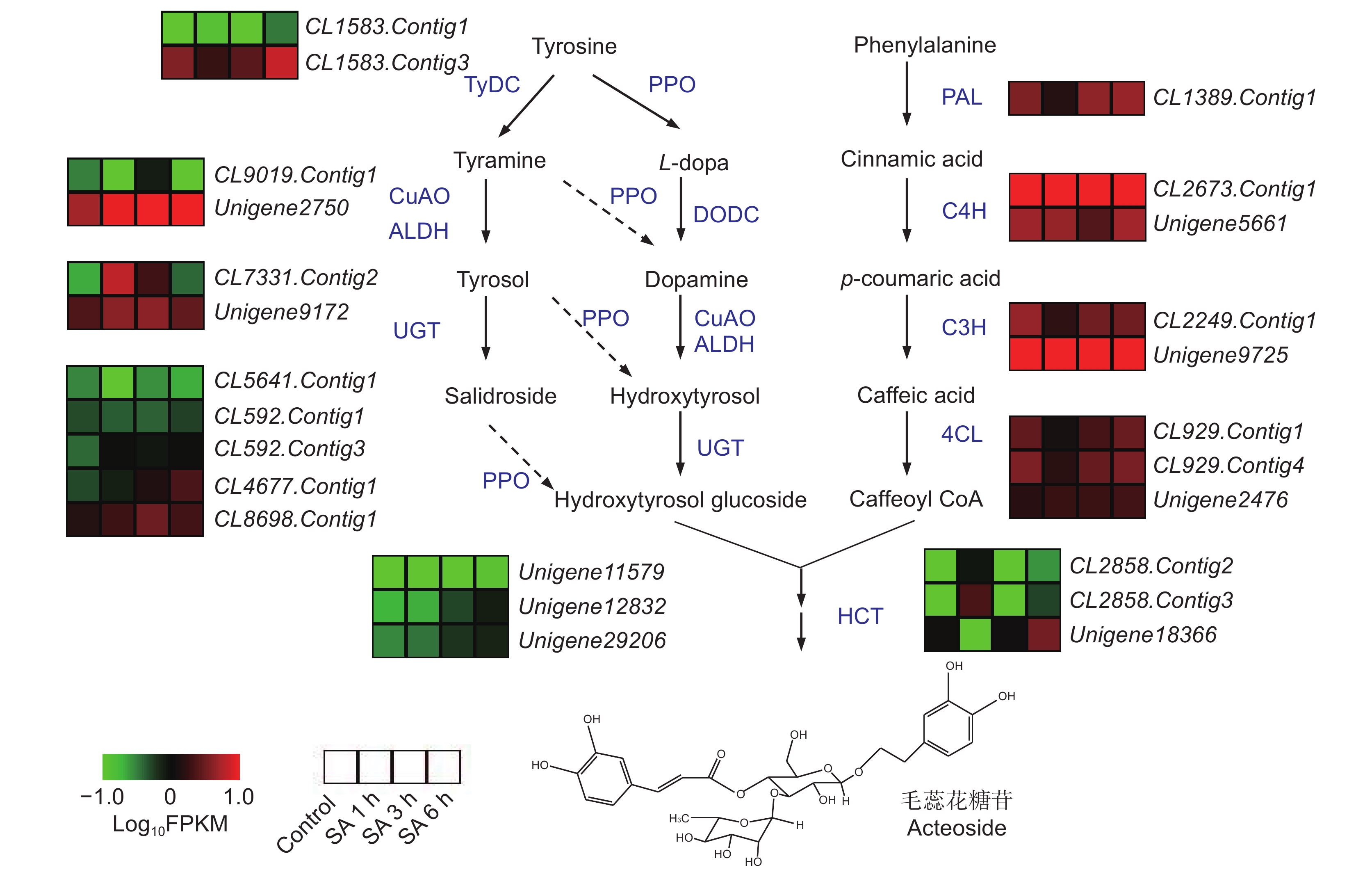
[4] 王丰青,杨旭,左鑫,苗春妍,张重义. 地黄全长转录组测序及苯乙醇苷合成途径催化酶基因鉴定[J]. 药学学报,2022,57(3):831−838. Wang FQ,Yang X,Zuo X,Miao CY,Zhang ZY. Full-length transcriptome sequence and identification of genes involved in phenylethanol glycoside biosynthesis in Rehmannia glutinosa[J]. Acta Pharmaceutica Sinica,2022,57 (3):831−838.
[5] 王丰青,王丽娜,智惊宇,张苗,杨超飞,等. 不同品种地黄中毛蕊花糖苷的动态积累规律变化[J]. 中国实验方剂学杂志,2017,23(24):78−83. doi: 10.13422/j.cnki.syfjx.2017240078 Wang FQ,Wang LN,Zhi JY,Zhang M,Yang CF,et al. Changes in dynamic accumulation of acteoside from different Rehmannia glutinosa cultivars[J]. Chinese Journal of Experimental Traditional Medical Formulae,2017,23 (24):78−83. doi: 10.13422/j.cnki.syfjx.2017240078
[6] 柴茂,董诚明,江道会,姚峰,王秀书. 不同品种怀地黄中梓醇和毛蕊花糖苷的高效液相色谱法测定[J]. 中医学报,2013,28(5):690−692. doi: 10.16368/j.issn.1674-8999.2013.05.050 Chai M,Dong CM,Jiang DH,Yao F,Wang XS. Determination of catalpol and verbascoside of different cultivars Rehmannia glutinosa Libosch. by high performance liquid chromatography[J]. China Journal of Chinese Medicine,2013,28 (5):690−692. doi: 10.16368/j.issn.1674-8999.2013.05.050
[7] 柴烨,郑立颖. 不同产地生地黄和熟地黄中毛蕊花糖苷含量的比较研究[J]. 甘肃科学学报,2015,27(3):37−40. doi: 10.16468/j.cnki.issn1004-0366.2015.03.010 Chai Y,Zheng LY. A comparative study of verbascoside content in Rehmannia root & prepared Rehmannia root from different places of production[J]. Journal of Gansu Sciences,2015,27 (3):37−40. doi: 10.16468/j.cnki.issn1004-0366.2015.03.010
[8] 王丰青,杨超飞,李铭铭,左鑫,杨旭,等. 密度对地黄生长及基因转录特性的影响分析[J]. 中国中药杂志,2021,46(17):4367−4379. doi: 10.19540/j.cnki.cjcmm.20210623.101 Wang FQ,Yang CF,Li MM,Zuo X,Yang X,et al. Effects of density on growth and gene transcription characteristics of Rehmannia glutinosa[J]. China Journal of Chinese Materia Medica,2021,46 (17):4367−4379. doi: 10.19540/j.cnki.cjcmm.20210623.101
[9] 王丰青,李欣容,杨超飞,智惊宇,张雪丽,等. 遮阴对地黄块根性状、光合特性及基因转录的影响[J]. 中草药,2019,50(18):4419−4429. doi: 10.7501/j.issn.0253-2670.2019.18.023 Wang FQ,Li XR,Yang CF,Zhi JY,Zhang XL,et al. Effects of shading on tuberous root traits,photosynthetic characteristics and gene transcription of Rehmannia glutinosa[J]. Chinese Traditional and Herbal Drugs,2019,50 (18):4419−4429. doi: 10.7501/j.issn.0253-2670.2019.18.023
[10] Wang FQ,Zhi JY,Zhang ZY,Wang LN,Suo YF,et al. Transcriptome analysis of salicylic acid treatment in Rehmannia glutinosa hairy roots using RNA-seq technique for identification of genes involved in acteoside biosynthesis[J]. Front Plant Sci,2017,8:787. doi: 10.3389/fpls.2017.00787
[11] Li MJ,Yang YH,Feng FJ,Zhang B,Chen SQ,et al. Differential proteomic analysis of replanted Rehmannia glutinosa roots by iTRAQ reveals molecular mechanisms for formation of replant disease[J]. BMC Plant Biol,2017,17 (1):116. doi: 10.1186/s12870-017-1060-0
[12] Ramirez-Estrada K,Vidal-Limon H,Hidalgo D,Moyano E,Golenioswki M,et al. Elicitation,an effective strategy for the biotechnological production of bioactive high-added value compounds in plant cell factories[J]. Molecules,2016,21 (2):182. doi: 10.3390/molecules21020182
[13] Yuan YJ,Wei ZJ,Miao ZQ,Wu JC. Acting paths of elicitors on Taxol biosynthesis pathway and their synergistic effect[J]. Biochem Eng J,2002,10 (2):77−83. doi: 10.1016/S1369-703X(01)00159-0
[14] Bae KH,Choi YE,Shin CG,Kim YY,Kim YS. Enhanced ginsenoside productivity by combination of ethephon and methyl jasmoante in ginseng (Panax ginseng C. A. Meyer) adventitious root cultures[J]. Biotechnol Lett,2006,28 (15):1163−1166. doi: 10.1007/s10529-006-9071-1
[15] Chen WH,Xu CM,Zeng JL,Zhao B,Wang XD,Wang YC. Improvement of echinacoside and acteoside production by two-stage elicitation in cell suspension culture of Cistanche deserticola[J]. World J Microbiol Biotechnol,2007,23 (10):1451−1458. doi: 10.1007/s11274-007-9389-4
[16] 王尧龙,黄璐琦,袁媛,查良平. 药用植物转录组研究进展[J]. 中国中药杂志,2015,40(11):2055−2061. Wang YL,Huang LQ,Yuan Y,Zha LP. Research advances on analysis of medicinal plants transcriptome[J]. China Journal of Chinese Materia Medica,2015,40 (11):2055−2061.
[17] Kumar D,Hazra S,Datta R,Chattopadhyay S. Transcriptome analysis of Arabidopsis mutants suggests a crosstalk between ABA,ethylene and GSH against combined cold and osmotic stress[J]. Sci Rep,2016,6:36867. doi: 10.1038/srep36867
[18] Li C,Tao RF,Li Y,Duan MH,Xu JH. Transcriptome analysis of the thermosensitive genic male-sterile line provides new insights into fertility alteration in rice (Oryza sativa)[J]. Genomics,2020,112 (3):2119−2129. doi: 10.1016/j.ygeno.2019.12.006
[19] Wu WZ,Yang HB,Xing P,Dong Y,Shen J,et al. Comparative transcriptome analysis revealed the freezing tolerance signaling events in winter rapeseed (Brassica rapa L. )[J]. Front Genet,2022,13:871825. doi: 10.3389/fgene.2022.871825
[20] Chang YJ,Wang MZ,Li J,Lu SF. Transcriptomic analysis reveals potential genes involved in tanshinone biosynthesis in Salvia miltiorrhiza[J]. Sci Rep,2019,9 (1):14929. doi: 10.1038/s41598-019-51535-9
[21] Zhao MZ,Lin YP,Wang YF,Li XY,Han YL,et al. Transcriptome analysis identifies strong candidate genes for ginsenoside biosynthesis and reveals its underlying molecular mechanism in Panax ginseng C. A. Meyer[J]. Sci Rep,2019,9 (1):615. doi: 10.1038/s41598-018-36349-5
[22] Li QQ,Liu CB,Huang CY,Wang MF,Long T,et al. Transcriptome and metabonomics analysis revealed the molecular mechanism of differential metabolite production of Dendrobium nobile under different epiphytic patterns[J]. Front Plant Sci,2022,13:868472. doi: 10.3389/fpls.2022.868472
[23] Ma LG,Dong CM,Song C,Wang XL,Zheng XK,et al. De novo genome assembly of the potent medicinal plant Rehmannia glutinosa using nanopore technology[J]. Comput Struct Biotechnol J,2021,19:3954−3963. doi: 10.1016/j.csbj.2021.07.006
[24] Saimaru H,Orihara Y. Biosynthesis of acteoside in cultured cells of Olea europaea[J]. J Nat Med,2010,64 (2):139−145. doi: 10.1007/s11418-009-0383-z
[25] 周延清,邵露营,郭萌萌,朱佳琳. 地黄C3H基因的克隆及生物信息学分析[J]. 广西植物,2020,40(9):1281−1287. doi: 10.11931/guihaia.gxzw201907060 Zhou YQ,Shao LY,Guo MM,Zhu JL. Cloning and bioinformatics analysis of C3H gene in Rehmannia glutinosa[J]. Guihaia,2020,40 (9):1281−1287. doi: 10.11931/guihaia.gxzw201907060
[26] 李欣容,智惊宇,杨超飞,李铭铭,左鑫,等. 地黄毛蕊花糖苷合酶基因的克隆、亚细胞定位与表达特性分析[J]. 中草药,2020,51(18):4739−4746. doi: 10.7501/j.issn.0253-2670.2020.18.018 Li XR,Zhi JY,Yang CF,Li MM,Zuo X,et al. Cloning,subcellular location and expression analysis of an acteoside synthase gene from Rehmannia glutinosa[J]. Chinese Traditional and Herbal Drugs,2020,51 (18):4739−4746. doi: 10.7501/j.issn.0253-2670.2020.18.018
[27] Yang YH,Yang MR,Zhu JY,Dong KW,Yi YJ,et al. Functional characterization of tyrosine decarboxylase genes that contribute to acteoside biosynthesis in Rehmannia glutinosa[J]. Planta,2022,255 (3):64. doi: 10.1007/s00425-022-03849-8
[28] Wang FQ,Li XR,Zuo X,Li MM,Miao CY,et al. Transcriptome-wide identification of WRKY transcription factor and functional characterization of RgWRKY37 involved in acteoside biosynthesis in Rehmannia glutinosa[J]. Front Plant Sci,2021,12:739853. doi: 10.3389/fpls.2021.739853
-
期刊类型引用(0)
其他类型引用(1)



 下载:
下载: